Aperçu
La principale caractéristique de TensorBoard est son interface graphique interactive. Cependant, les utilisateurs veulent parfois lire les journaux de programmation données stockées dans TensorBoard, à des fins telles que la réalisation d' analyses post-hoc et la création d' Visualisations personnalisées des données du journal.
TensorBoard 2.3 supporte ce cas d'utilisation avec tensorboard.data.experimental.ExperimentFromDev() . Il permet un accès programmatique de TensorBoard journaux scalaires . Cette page montre l'utilisation de base de cette nouvelle API.
Installer
Pour utiliser l'API de programmation, assurez-vous d' installer pandas à côté tensorboard .
Nous allons utiliser matplotlib et seaborn pour les tracés personnalisés dans ce guide, mais vous pouvez choisir votre outil préféré pour analyser et visualiser DataFrame s.
pip install tensorboard pandaspip install matplotlib seaborn
from packaging import version
import pandas as pd
from matplotlib import pyplot as plt
import seaborn as sns
from scipy import stats
import tensorboard as tb
major_ver, minor_ver, _ = version.parse(tb.__version__).release
assert major_ver >= 2 and minor_ver >= 3, \
"This notebook requires TensorBoard 2.3 or later."
print("TensorBoard version: ", tb.__version__)
TensorBoard version: 2.3.0a20200626
Chargement scalaires TensorBoard comme pandas.DataFrame
Une fois qu'un logdir de TensorBoard a été transféré à TensorBoard.dev, il devient ce que nous appelons une expérience. Chaque expérience a un identifiant unique, qui peut être trouvé dans l'URL TensorBoard.dev de l'expérience. Pour en dessous de notre démonstration, nous utiliserons une expérience TensorBoard.dev à: https://tensorboard.dev/experiment/c1KCv3X3QvGwaXfgX1c4tg
experiment_id = "c1KCv3X3QvGwaXfgX1c4tg"
experiment = tb.data.experimental.ExperimentFromDev(experiment_id)
df = experiment.get_scalars()
df
df est un pandas.DataFrame qui contient tous les journaux scalaires de l'expérience.
Les colonnes du DataFrame sont:
-
run: chaque essai correspond à un sous - répertoire du logdir d' origine. Dans cette expérience, chaque exécution provient d'un entraînement complet d'un réseau de neurones convolutifs (CNN) sur l'ensemble de données MNIST avec un type d'optimiseur donné (un hyperparamètre d'entraînement). CetteDataFramedeDataFramecontient plusieurs séries telles, qui correspondent à des cycles de formation répétées sous différents types d'optimisation. -
tag: cela décrit ce que lavaluedans le même moyen de ligne, qui est, ce que la valeur métrique représente la ligne. Dans cette expérience, nous avons seulement deux balises uniques:epoch_accuracyetepoch_losspour les mesures de précision et de perte respectivement. -
step: Ceci est un nombre qui reflète l'ordre de série de la rangée correspondant à sa course. Icistepfait référence au numéro d'époque. Si vous souhaitez obtenir les horodatages en plus desstepvaleurs, vous pouvez utiliser l'argument mot - cléinclude_wall_time=Truelorsque vous appelezget_scalars(). -
value: Ceci est la valeur numérique réelle d'intérêt. Comme décrit ci - dessus, chaquevaluedans ce cas particulierDataFramedetagDataFrameest soit une perte ou une précision, en fonction de l'tagde la rangée.
print(df["run"].unique())
print(df["tag"].unique())
['adam,run_1/train' 'adam,run_1/validation' 'adam,run_2/train' 'adam,run_2/validation' 'adam,run_3/train' 'adam,run_3/validation' 'adam,run_4/train' 'adam,run_4/validation' 'adam,run_5/train' 'adam,run_5/validation' 'rmsprop,run_1/train' 'rmsprop,run_1/validation' 'rmsprop,run_2/train' 'rmsprop,run_2/validation' 'rmsprop,run_3/train' 'rmsprop,run_3/validation' 'rmsprop,run_4/train' 'rmsprop,run_4/validation' 'rmsprop,run_5/train' 'rmsprop,run_5/validation' 'sgd,run_1/train' 'sgd,run_1/validation' 'sgd,run_2/train' 'sgd,run_2/validation' 'sgd,run_3/train' 'sgd,run_3/validation' 'sgd,run_4/train' 'sgd,run_4/validation' 'sgd,run_5/train' 'sgd,run_5/validation'] ['epoch_accuracy' 'epoch_loss']
Obtenir un DataFrame pivoté (format large)
Dans notre expérience, les deux balises ( epoch_loss et epoch_accuracy ) sont présents dans le même ensemble d'étapes dans chaque course. Cela permet d'obtenir un « grand forme » DataFrame directement à partir get_scalars() en utilisant le pivot=True argument mot - clé. La grande forme DataFrame a toutes ses balises incluses sous forme de colonnes du dataframe, qui est plus commode de travailler avec dans certains cas , dont celui - ci.
Toutefois, méfiez - vous que si la condition d'avoir des ensembles de valeurs de pas uniformes dans tous les tags dans tous les essais n'est pas remplie, à l' aide pivot=True entraînera une erreur.
dfw = experiment.get_scalars(pivot=True)
dfw
Notez qu'au lieu d'une seule colonne « valeur », la grande forme dataframe comprend les deux balises (métriques) comme ses colonnes explicitement: epoch_accuracy et epoch_loss .
Enregistrement du DataFrame au format CSV
pandas.DataFrame a une bonne interopérabilité avec CSV . Vous pouvez le stocker en tant que fichier CSV local et le recharger ultérieurement. Par exemple:
csv_path = '/tmp/tb_experiment_1.csv'
dfw.to_csv(csv_path, index=False)
dfw_roundtrip = pd.read_csv(csv_path)
pd.testing.assert_frame_equal(dfw_roundtrip, dfw)
Effectuer une visualisation personnalisée et une analyse statistique
# Filter the DataFrame to only validation data, which is what the subsequent
# analyses and visualization will be focused on.
dfw_validation = dfw[dfw.run.str.endswith("/validation")]
# Get the optimizer value for each row of the validation DataFrame.
optimizer_validation = dfw_validation.run.apply(lambda run: run.split(",")[0])
plt.figure(figsize=(16, 6))
plt.subplot(1, 2, 1)
sns.lineplot(data=dfw_validation, x="step", y="epoch_accuracy",
hue=optimizer_validation).set_title("accuracy")
plt.subplot(1, 2, 2)
sns.lineplot(data=dfw_validation, x="step", y="epoch_loss",
hue=optimizer_validation).set_title("loss")
Text(0.5, 1.0, 'loss')
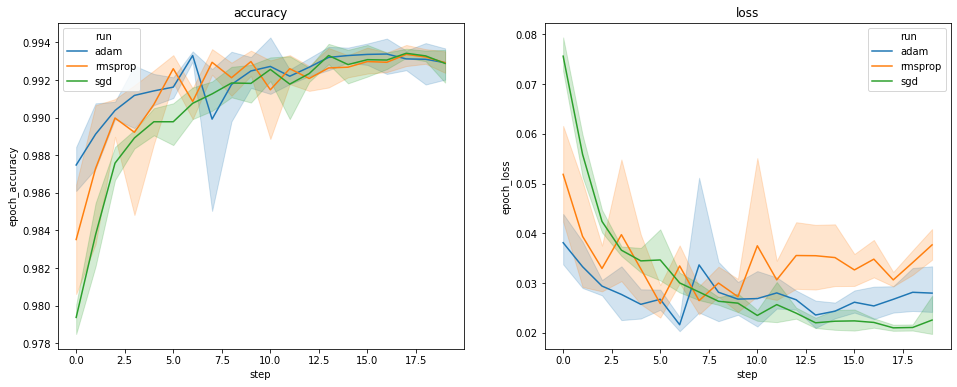
Les graphiques ci-dessus montrent les évolutions temporelles de la précision de la validation et de la perte de validation. Chaque courbe montre la moyenne sur 5 exécutions sous un type d'optimiseur. Merci à une fonctionnalité intégrée de seaborn.lineplot() , chaque courbe affiche également de 1 écart - type autour de la moyenne, ce qui nous donne une idée claire de la variabilité de ces courbes et l'importance des différences entre les trois types d'optimisation. Cette visualisation de la variabilité n'est pas encore prise en charge dans l'interface graphique de TensorBoard.
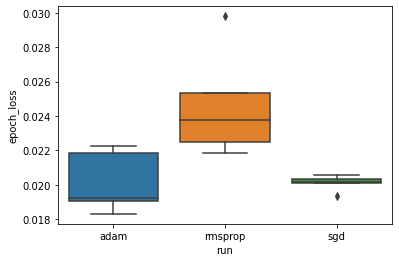
Nous voulons étudier l'hypothèse que la perte de validation minimale diffère significativement entre les optimiseurs "adam", "rmsprop" et "sgd". Nous extrayons donc un DataFrame pour la perte de validation minimale sous chacun des optimiseurs.
Ensuite, nous réalisons un boxplot pour visualiser la différence dans les pertes de validation minimales.
adam_min_val_loss = dfw_validation.loc[optimizer_validation=="adam", :].groupby(
"run", as_index=False).agg({"epoch_loss": "min"})
rmsprop_min_val_loss = dfw_validation.loc[optimizer_validation=="rmsprop", :].groupby(
"run", as_index=False).agg({"epoch_loss": "min"})
sgd_min_val_loss = dfw_validation.loc[optimizer_validation=="sgd", :].groupby(
"run", as_index=False).agg({"epoch_loss": "min"})
min_val_loss = pd.concat([adam_min_val_loss, rmsprop_min_val_loss, sgd_min_val_loss])
sns.boxplot(data=min_val_loss, y="epoch_loss",
x=min_val_loss.run.apply(lambda run: run.split(",")[0]))
<matplotlib.axes._subplots.AxesSubplot at 0x7f5e017c8150>
# Perform pairwise comparisons between the minimum validation losses
# from the three optimizers.
_, p_adam_vs_rmsprop = stats.ttest_ind(
adam_min_val_loss["epoch_loss"],
rmsprop_min_val_loss["epoch_loss"])
_, p_adam_vs_sgd = stats.ttest_ind(
adam_min_val_loss["epoch_loss"],
sgd_min_val_loss["epoch_loss"])
_, p_rmsprop_vs_sgd = stats.ttest_ind(
rmsprop_min_val_loss["epoch_loss"],
sgd_min_val_loss["epoch_loss"])
print("adam vs. rmsprop: p = %.4f" % p_adam_vs_rmsprop)
print("adam vs. sgd: p = %.4f" % p_adam_vs_sgd)
print("rmsprop vs. sgd: p = %.4f" % p_rmsprop_vs_sgd)
adam vs. rmsprop: p = 0.0244 adam vs. sgd: p = 0.9749 rmsprop vs. sgd: p = 0.0135
Par conséquent, à un niveau de signification de 0,05, notre analyse confirme notre hypothèse selon laquelle la perte de validation minimale est significativement plus élevée (c'est-à-dire pire) dans l'optimiseur rmsprop par rapport aux deux autres optimiseurs inclus dans notre expérience.
En résumé, ce tutoriel donne un exemple de la façon d'accéder aux données scalaires comme panda.DataFrame s de TensorBoard.dev. Il démontre le genre d'analyses souples et puissantes et la visualisation que vous pouvez faire avec le DataFrame s.
