 Ver no TensorFlow.org Ver no TensorFlow.org |  Executar no Google Colab Executar no Google Colab |  Ver fonte no GitHub Ver fonte no GitHub |  Baixar caderno Baixar caderno |
Probabilística análise de componentes principais (PCA) é uma técnica de redução dimensionalidade que analisa os dados através de um espaço dimensional inferior latente ( Tipping e Bishop 1999 ). É frequentemente usado quando há valores ausentes nos dados ou para dimensionamento multidimensional.
Importações
import functools
import warnings
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import tensorflow.compat.v2 as tf
import tensorflow_probability as tfp
from tensorflow_probability import bijectors as tfb
from tensorflow_probability import distributions as tfd
tf.enable_v2_behavior()
plt.style.use("ggplot")
warnings.filterwarnings('ignore')
O Modelo
Considere-se um conjunto de dados \(\mathbf{X} = \{\mathbf{x}_n\}\) de \(N\) pontos de dados, em que cada ponto de dados é \(D\)-dimensional, $ \ mathbf {x} _n \ in \ mathbb {R} ^ D\(. We aim to represent each \)\ mathbf {x} _n $ sob uma variável latente \(\mathbf{z}_n \in \mathbb{R}^K\) com dimensão inferior, $ K <D\(. The set of principal axes \)\ mathbf {W} $ refere-se as variáveis latentes para os dados.
Especificamente, assumimos que cada variável latente é normalmente distribuída,
\[ \begin{equation*} \mathbf{z}_n \sim N(\mathbf{0}, \mathbf{I}). \end{equation*} \]
O ponto de dados correspondente é gerado por meio de uma projeção,
\[ \begin{equation*} \mathbf{x}_n \mid \mathbf{z}_n \sim N(\mathbf{W}\mathbf{z}_n, \sigma^2\mathbf{I}), \end{equation*} \]
onde a matriz \(\mathbf{W}\in\mathbb{R}^{D\times K}\) são conhecidos como os eixos principais. Em PCA probabilística, estamos normalmente interessados em estimar os eixos principais \(\mathbf{W}\) eo termo de ruído\(\sigma^2\).
A PCA probabilística generaliza a PCA clássica. Marginalizando a variável latente, a distribuição de cada ponto de dados é
\[ \begin{equation*} \mathbf{x}_n \sim N(\mathbf{0}, \mathbf{W}\mathbf{W}^\top + \sigma^2\mathbf{I}). \end{equation*} \]
PCA clássico é o caso específico do PCA probabilística quando a covariância do ruído torna-se infinitamente pequeno, \(\sigma^2 \to 0\).
Montamos nosso modelo abaixo. Em nossa análise, assumimos \(\sigma\) é conhecido, e em vez de ponto de estimar \(\mathbf{W}\) como um parâmetro modelo, coloque uma prévia sobre ele, a fim de inferir uma distribuição mais eixos principais. Vamos expressar o modelo como um TFP JointDistribution, especificamente, vamos usar JointDistributionCoroutineAutoBatched .
def probabilistic_pca(data_dim, latent_dim, num_datapoints, stddv_datapoints):
w = yield tfd.Normal(loc=tf.zeros([data_dim, latent_dim]),
scale=2.0 * tf.ones([data_dim, latent_dim]),
name="w")
z = yield tfd.Normal(loc=tf.zeros([latent_dim, num_datapoints]),
scale=tf.ones([latent_dim, num_datapoints]),
name="z")
x = yield tfd.Normal(loc=tf.matmul(w, z),
scale=stddv_datapoints,
name="x")
num_datapoints = 5000
data_dim = 2
latent_dim = 1
stddv_datapoints = 0.5
concrete_ppca_model = functools.partial(probabilistic_pca,
data_dim=data_dim,
latent_dim=latent_dim,
num_datapoints=num_datapoints,
stddv_datapoints=stddv_datapoints)
model = tfd.JointDistributionCoroutineAutoBatched(concrete_ppca_model)
Os dados
Podemos usar o modelo para gerar dados por amostragem da distribuição prévia conjunta.
actual_w, actual_z, x_train = model.sample()
print("Principal axes:")
print(actual_w)
Principal axes: tf.Tensor( [[ 2.2801023] [-1.1619819]], shape=(2, 1), dtype=float32)
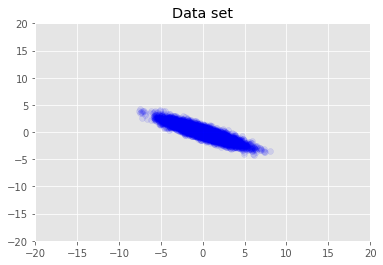
Visualizamos o conjunto de dados.
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1)
plt.axis([-20, 20, -20, 20])
plt.title("Data set")
plt.show()
Máxima Inferência Posterior
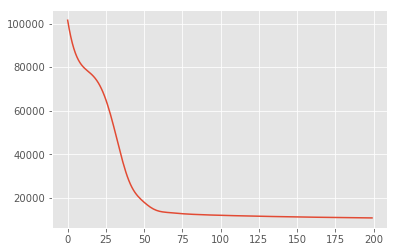
Buscamos primeiro a estimativa pontual de variáveis latentes que maximize a densidade de probabilidade posterior. Isto é conhecido como um máximo inferência (MAP) a posteriori, e é feito por meio do cálculo dos valores de \(\mathbf{W}\) e \(\mathbf{Z}\) que maximizam a densidade posterior \(p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X}) \propto p(\mathbf{W}, \mathbf{Z}, \mathbf{X})\).
w = tf.Variable(tf.random.normal([data_dim, latent_dim]))
z = tf.Variable(tf.random.normal([latent_dim, num_datapoints]))
target_log_prob_fn = lambda w, z: model.log_prob((w, z, x_train))
losses = tfp.math.minimize(
lambda: -target_log_prob_fn(w, z),
optimizer=tf.optimizers.Adam(learning_rate=0.05),
num_steps=200)
plt.plot(losses)
[<matplotlib.lines.Line2D at 0x7f19897a42e8>]
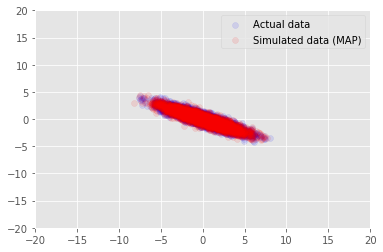
Nós podemos usar o modelo para dados de exemplo para os valores inferidos para \(\mathbf{W}\) e \(\mathbf{Z}\), e comparar com o conjunto de dados real que condicionando a.
print("MAP-estimated axes:")
print(w)
_, _, x_generated = model.sample(value=(w, z, None))
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1, label='Actual data')
plt.scatter(x_generated[0, :], x_generated[1, :], color='red', alpha=0.1, label='Simulated data (MAP)')
plt.legend()
plt.axis([-20, 20, -20, 20])
plt.show()
MAP-estimated axes:
<tf.Variable 'Variable:0' shape=(2, 1) dtype=float32, numpy=
array([[ 2.9135954],
[-1.4826864]], dtype=float32)>
Inferência Variacional
O MAP pode ser usado para encontrar a moda (ou uma das modas) da distribuição a posteriori, mas não fornece outras informações sobre ela. A seguir, utilizar inferência variacional, onde o distribtion posterior \(p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X})\) é aproximada usando uma distribuição variacional \(q(\mathbf{W}, \mathbf{Z})\) parametrizado pelo \(\boldsymbol{\lambda}\). O objectivo é encontrar os parâmetros variacional \(\boldsymbol{\lambda}\) que minimizar a divergência KL entre q e posterior, \(\mathrm{KL}(q(\mathbf{W}, \mathbf{Z}) \mid\mid p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X}))\), ou equivalentemente, que maximize a evidência limite inferior, \(\mathbb{E}_{q(\mathbf{W},\mathbf{Z};\boldsymbol{\lambda})}\left[ \log p(\mathbf{W},\mathbf{Z},\mathbf{X}) - \log q(\mathbf{W},\mathbf{Z}; \boldsymbol{\lambda}) \right]\).
qw_mean = tf.Variable(tf.random.normal([data_dim, latent_dim]))
qz_mean = tf.Variable(tf.random.normal([latent_dim, num_datapoints]))
qw_stddv = tfp.util.TransformedVariable(1e-4 * tf.ones([data_dim, latent_dim]),
bijector=tfb.Softplus())
qz_stddv = tfp.util.TransformedVariable(
1e-4 * tf.ones([latent_dim, num_datapoints]),
bijector=tfb.Softplus())
def factored_normal_variational_model():
qw = yield tfd.Normal(loc=qw_mean, scale=qw_stddv, name="qw")
qz = yield tfd.Normal(loc=qz_mean, scale=qz_stddv, name="qz")
surrogate_posterior = tfd.JointDistributionCoroutineAutoBatched(
factored_normal_variational_model)
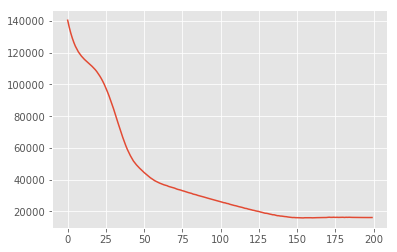
losses = tfp.vi.fit_surrogate_posterior(
target_log_prob_fn,
surrogate_posterior=surrogate_posterior,
optimizer=tf.optimizers.Adam(learning_rate=0.05),
num_steps=200)
print("Inferred axes:")
print(qw_mean)
print("Standard Deviation:")
print(qw_stddv)
plt.plot(losses)
plt.show()
Inferred axes:
<tf.Variable 'Variable:0' shape=(2, 1) dtype=float32, numpy=
array([[ 2.4168603],
[-1.2236133]], dtype=float32)>
Standard Deviation:
<TransformedVariable: dtype=float32, shape=[2, 1], fn="softplus", numpy=
array([[0.0042499 ],
[0.00598824]], dtype=float32)>
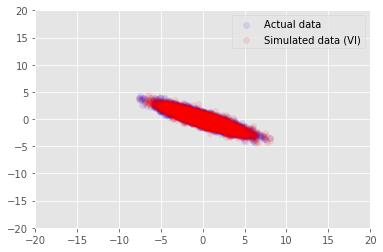
posterior_samples = surrogate_posterior.sample(50)
_, _, x_generated = model.sample(value=(posterior_samples))
# It's a pain to plot all 5000 points for each of our 50 posterior samples, so
# let's subsample to get the gist of the distribution.
x_generated = tf.reshape(tf.transpose(x_generated, [1, 0, 2]), (2, -1))[:, ::47]
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1, label='Actual data')
plt.scatter(x_generated[0, :], x_generated[1, :], color='red', alpha=0.1, label='Simulated data (VI)')
plt.legend()
plt.axis([-20, 20, -20, 20])
plt.show()
Reconhecimentos
Este tutorial foi escrito originalmente em Edward 1,0 ( fonte ). Agradecemos a todos os colaboradores que escreveram e revisaram essa versão.
Referências
[1]: Michael E. Tipping e Christopher M. Bishop. Análise probabilística de componentes principais. Journal of Royal Statistical Society: Série B (Metodologia Estatística), 61 (3): 611-622, 1999.
