 TensorFlow.org पर देखें TensorFlow.org पर देखें |  Google Colab में चलाएं Google Colab में चलाएं |  गिटहब पर देखें गिटहब पर देखें |  नोटबुक डाउनलोड करें नोटबुक डाउनलोड करें |  टीएफ हब मॉडल देखें टीएफ हब मॉडल देखें |
CORD-19 फिरकी पाठ TF-हब से मॉड्यूल embedding ( https://tfhub.dev/tensorflow/cord-19/swivel-128d/3 ) प्राकृतिक भाषाओं COVID -19 से संबंधित पाठ का विश्लेषण समर्थन शोधकर्ताओं के लिए बनाया गया था। ये embeddings शीर्षक, लेखक, सार, शरीर पाठ, और संदर्भ में लेख के शीर्षक पर प्रशिक्षित किया गया CORD-19 डाटासेट ।
इस कोलाब में हम करेंगे:
- एम्बेडिंग स्पेस में शब्दार्थ समान शब्दों का विश्लेषण करें
- CORD-19 एम्बेडिंग का उपयोग करके SciCite डेटासेट पर क्लासिफायरियर को प्रशिक्षित करें
सेट अप
import functools
import itertools
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import pandas as pd
import tensorflow as tf
import tensorflow_datasets as tfds
import tensorflow_hub as hub
from tqdm import trange
एम्बेडिंग का विश्लेषण करें
आइए विभिन्न शब्दों के बीच सहसंबंध मैट्रिक्स की गणना और प्लॉटिंग करके एम्बेडिंग का विश्लेषण करके शुरू करें। यदि एम्बेडिंग ने अलग-अलग शब्दों के अर्थ को सफलतापूर्वक पकड़ना सीख लिया है, तो शब्दार्थ समान शब्दों के एम्बेडिंग वैक्टर एक साथ होने चाहिए। आइए कुछ COVID-19 संबंधित शर्तों पर एक नज़र डालें।
# Use the inner product between two embedding vectors as the similarity measure
def plot_correlation(labels, features):
corr = np.inner(features, features)
corr /= np.max(corr)
sns.heatmap(corr, xticklabels=labels, yticklabels=labels)
# Generate embeddings for some terms
queries = [
# Related viruses
'coronavirus', 'SARS', 'MERS',
# Regions
'Italy', 'Spain', 'Europe',
# Symptoms
'cough', 'fever', 'throat'
]
module = hub.load('https://tfhub.dev/tensorflow/cord-19/swivel-128d/3')
embeddings = module(queries)
plot_correlation(queries, embeddings)

हम देख सकते हैं कि एम्बेडिंग ने विभिन्न शब्दों के अर्थ को सफलतापूर्वक पकड़ लिया है। प्रत्येक शब्द अपने क्लस्टर के अन्य शब्दों के समान है (अर्थात "कोरोनावायरस" "SARS" और "MERS" से अत्यधिक संबंधित है), जबकि वे अन्य समूहों की शर्तों से भिन्न हैं (अर्थात "SARS" और "स्पेन" के बीच समानता है) 0 के करीब)।
अब देखते हैं कि हम किसी विशिष्ट कार्य को हल करने के लिए इन एम्बेडिंग का उपयोग कैसे कर सकते हैं।
SciCite: प्रशस्ति पत्र आशय वर्गीकरण
यह खंड दिखाता है कि टेक्स्ट वर्गीकरण जैसे डाउनस्ट्रीम कार्यों के लिए कोई एम्बेडिंग का उपयोग कैसे कर सकता है। हम इस्तेमाल करेंगे SciCite डाटासेट शैक्षिक पेपर में वर्गीकृत प्रशस्ति पत्र उद्देश्यों के लिए TensorFlow डेटासेट से। एक अकादमिक पेपर से उद्धरण के साथ एक वाक्य को देखते हुए, वर्गीकृत करें कि क्या उद्धरण का मुख्य उद्देश्य पृष्ठभूमि की जानकारी, विधियों का उपयोग, या परिणामों की तुलना करना है।
builder = tfds.builder(name='scicite')
builder.download_and_prepare()
train_data, validation_data, test_data = builder.as_dataset(
split=('train', 'validation', 'test'),
as_supervised=True)
आइए प्रशिक्षण सेट से कुछ लेबल किए गए उदाहरणों पर एक नज़र डालें
NUM_EXAMPLES = 10
TEXT_FEATURE_NAME = builder.info.supervised_keys[0]
LABEL_NAME = builder.info.supervised_keys[1]
def label2str(numeric_label):
m = builder.info.features[LABEL_NAME].names
return m[numeric_label]
data = next(iter(train_data.batch(NUM_EXAMPLES)))
pd.DataFrame({
TEXT_FEATURE_NAME: [ex.numpy().decode('utf8') for ex in data[0]],
LABEL_NAME: [label2str(x) for x in data[1]]
})
सिटन इंटेंट क्लासिफायरियर का प्रशिक्षण
हम पर एक वर्गीकारक को प्रशिक्षित करेंगे SciCite डाटासेट Keras का उपयोग कर। आइए एक मॉडल बनाते हैं जो शीर्ष पर वर्गीकरण परत के साथ CORD-19 एम्बेडिंग का उपयोग करता है।
हाइपरपैरामीटर
EMBEDDING = 'https://tfhub.dev/tensorflow/cord-19/swivel-128d/3'
TRAINABLE_MODULE = False
hub_layer = hub.KerasLayer(EMBEDDING, input_shape=[],
dtype=tf.string, trainable=TRAINABLE_MODULE)
model = tf.keras.Sequential()
model.add(hub_layer)
model.add(tf.keras.layers.Dense(3))
model.summary()
model.compile(optimizer='adam',
loss=tf.keras.losses.SparseCategoricalCrossentropy(from_logits=True),
metrics=['accuracy'])
Model: "sequential"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
keras_layer (KerasLayer) (None, 128) 17301632
dense (Dense) (None, 3) 387
=================================================================
Total params: 17,302,019
Trainable params: 387
Non-trainable params: 17,301,632
_________________________________________________________________
मॉडल को प्रशिक्षित और मूल्यांकन करें
आइए SciCite कार्य पर प्रदर्शन देखने के लिए मॉडल को प्रशिक्षित और मूल्यांकन करें
EPOCHS = 35
BATCH_SIZE = 32
history = model.fit(train_data.shuffle(10000).batch(BATCH_SIZE),
epochs=EPOCHS,
validation_data=validation_data.batch(BATCH_SIZE),
verbose=1)
Epoch 1/35 257/257 [==============================] - 3s 7ms/step - loss: 0.9244 - accuracy: 0.5924 - val_loss: 0.7915 - val_accuracy: 0.6627 Epoch 2/35 257/257 [==============================] - 2s 5ms/step - loss: 0.7097 - accuracy: 0.7152 - val_loss: 0.6799 - val_accuracy: 0.7358 Epoch 3/35 257/257 [==============================] - 2s 7ms/step - loss: 0.6317 - accuracy: 0.7551 - val_loss: 0.6285 - val_accuracy: 0.7544 Epoch 4/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5938 - accuracy: 0.7687 - val_loss: 0.6032 - val_accuracy: 0.7566 Epoch 5/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5724 - accuracy: 0.7750 - val_loss: 0.5871 - val_accuracy: 0.7653 Epoch 6/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5580 - accuracy: 0.7825 - val_loss: 0.5800 - val_accuracy: 0.7653 Epoch 7/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5484 - accuracy: 0.7870 - val_loss: 0.5711 - val_accuracy: 0.7718 Epoch 8/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5417 - accuracy: 0.7896 - val_loss: 0.5648 - val_accuracy: 0.7806 Epoch 9/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5356 - accuracy: 0.7902 - val_loss: 0.5628 - val_accuracy: 0.7740 Epoch 10/35 257/257 [==============================] - 2s 7ms/step - loss: 0.5313 - accuracy: 0.7903 - val_loss: 0.5581 - val_accuracy: 0.7849 Epoch 11/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5277 - accuracy: 0.7928 - val_loss: 0.5555 - val_accuracy: 0.7838 Epoch 12/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5242 - accuracy: 0.7940 - val_loss: 0.5528 - val_accuracy: 0.7849 Epoch 13/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5215 - accuracy: 0.7947 - val_loss: 0.5522 - val_accuracy: 0.7828 Epoch 14/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5190 - accuracy: 0.7961 - val_loss: 0.5527 - val_accuracy: 0.7751 Epoch 15/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5176 - accuracy: 0.7940 - val_loss: 0.5492 - val_accuracy: 0.7806 Epoch 16/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5154 - accuracy: 0.7978 - val_loss: 0.5500 - val_accuracy: 0.7817 Epoch 17/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5136 - accuracy: 0.7968 - val_loss: 0.5488 - val_accuracy: 0.7795 Epoch 18/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5127 - accuracy: 0.7967 - val_loss: 0.5504 - val_accuracy: 0.7838 Epoch 19/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5111 - accuracy: 0.7970 - val_loss: 0.5470 - val_accuracy: 0.7860 Epoch 20/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5101 - accuracy: 0.7972 - val_loss: 0.5471 - val_accuracy: 0.7871 Epoch 21/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5082 - accuracy: 0.7997 - val_loss: 0.5483 - val_accuracy: 0.7784 Epoch 22/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5077 - accuracy: 0.7995 - val_loss: 0.5471 - val_accuracy: 0.7860 Epoch 23/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5064 - accuracy: 0.8012 - val_loss: 0.5439 - val_accuracy: 0.7871 Epoch 24/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5057 - accuracy: 0.7990 - val_loss: 0.5476 - val_accuracy: 0.7882 Epoch 25/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5050 - accuracy: 0.7996 - val_loss: 0.5442 - val_accuracy: 0.7937 Epoch 26/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5045 - accuracy: 0.7999 - val_loss: 0.5455 - val_accuracy: 0.7860 Epoch 27/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5032 - accuracy: 0.7991 - val_loss: 0.5435 - val_accuracy: 0.7893 Epoch 28/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5034 - accuracy: 0.8022 - val_loss: 0.5431 - val_accuracy: 0.7882 Epoch 29/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5025 - accuracy: 0.8017 - val_loss: 0.5441 - val_accuracy: 0.7937 Epoch 30/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5017 - accuracy: 0.8013 - val_loss: 0.5463 - val_accuracy: 0.7838 Epoch 31/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5015 - accuracy: 0.8017 - val_loss: 0.5453 - val_accuracy: 0.7871 Epoch 32/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5011 - accuracy: 0.8014 - val_loss: 0.5448 - val_accuracy: 0.7915 Epoch 33/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5006 - accuracy: 0.8025 - val_loss: 0.5432 - val_accuracy: 0.7893 Epoch 34/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5005 - accuracy: 0.8008 - val_loss: 0.5448 - val_accuracy: 0.7904 Epoch 35/35 257/257 [==============================] - 2s 5ms/step - loss: 0.4996 - accuracy: 0.8016 - val_loss: 0.5448 - val_accuracy: 0.7915
from matplotlib import pyplot as plt
def display_training_curves(training, validation, title, subplot):
if subplot%10==1: # set up the subplots on the first call
plt.subplots(figsize=(10,10), facecolor='#F0F0F0')
plt.tight_layout()
ax = plt.subplot(subplot)
ax.set_facecolor('#F8F8F8')
ax.plot(training)
ax.plot(validation)
ax.set_title('model '+ title)
ax.set_ylabel(title)
ax.set_xlabel('epoch')
ax.legend(['train', 'valid.'])
display_training_curves(history.history['accuracy'], history.history['val_accuracy'], 'accuracy', 211)
display_training_curves(history.history['loss'], history.history['val_loss'], 'loss', 212)
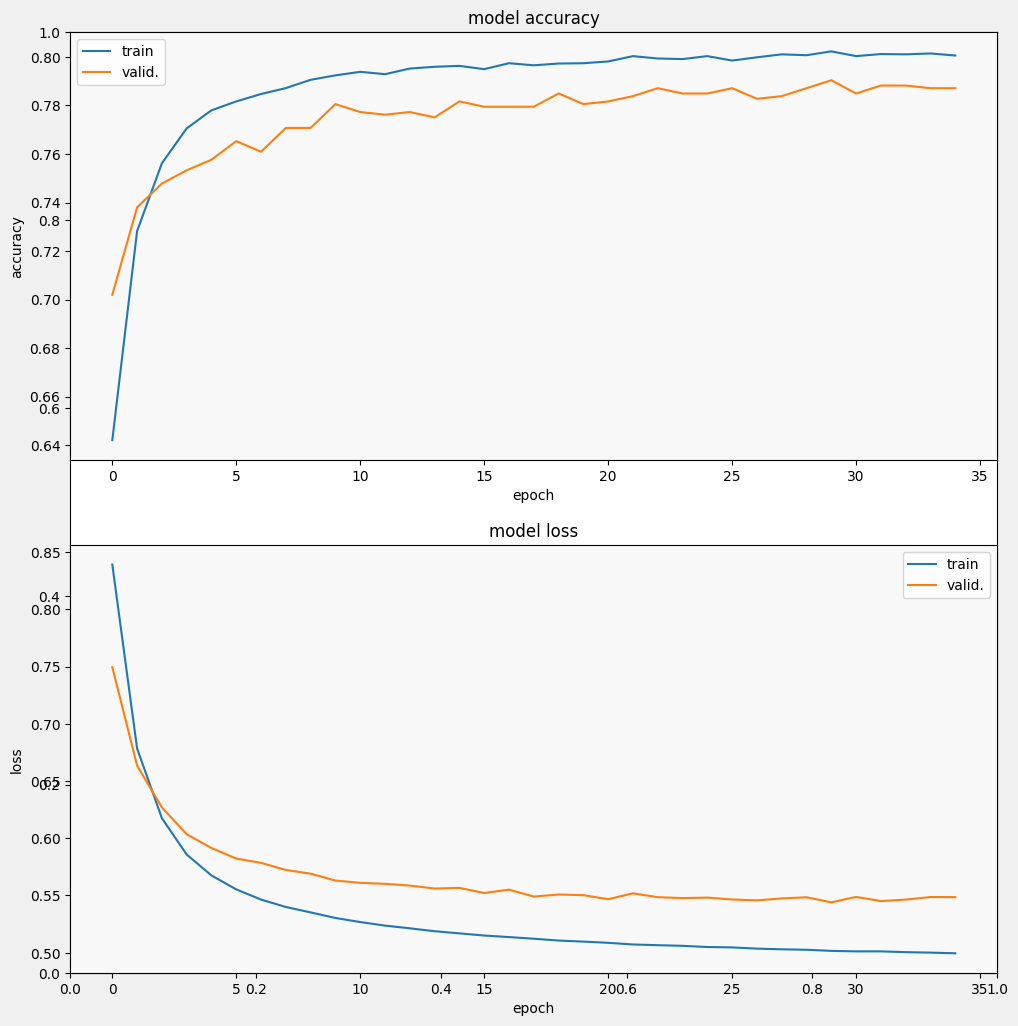
मॉडल का मूल्यांकन करें
और देखते हैं कि मॉडल कैसा प्रदर्शन करता है। दो मान लौटाए जाएंगे। हानि (एक संख्या जो हमारी त्रुटि का प्रतिनिधित्व करती है, कम मान बेहतर होते हैं), और सटीकता।
results = model.evaluate(test_data.batch(512), verbose=2)
for name, value in zip(model.metrics_names, results):
print('%s: %.3f' % (name, value))
4/4 - 0s - loss: 0.5357 - accuracy: 0.7891 - 441ms/epoch - 110ms/step loss: 0.536 accuracy: 0.789
हम देख सकते हैं कि नुकसान तेजी से घटता है, जबकि विशेष रूप से सटीकता तेजी से बढ़ती है। आइए यह जांचने के लिए कुछ उदाहरण देखें कि भविष्यवाणी सही लेबल से कैसे संबंधित है:
prediction_dataset = next(iter(test_data.batch(20)))
prediction_texts = [ex.numpy().decode('utf8') for ex in prediction_dataset[0]]
prediction_labels = [label2str(x) for x in prediction_dataset[1]]
predictions = [
label2str(x) for x in np.argmax(model.predict(prediction_texts), axis=-1)]
pd.DataFrame({
TEXT_FEATURE_NAME: prediction_texts,
LABEL_NAME: prediction_labels,
'prediction': predictions
})
हम देख सकते हैं कि इस यादृच्छिक नमूने के लिए, मॉडल ज्यादातर बार सही लेबल की भविष्यवाणी करता है, यह दर्शाता है कि यह वैज्ञानिक वाक्यों को अच्छी तरह से एम्बेड कर सकता है।
आगे क्या होगा?
अब जब आप TF-Hub से CORD-19 स्विवेल एम्बेडिंग के बारे में कुछ और जान गए हैं, तो हम आपको COVID-19 संबंधित शैक्षणिक ग्रंथों से वैज्ञानिक अंतर्दृष्टि प्राप्त करने में योगदान करने के लिए CORD-19 कागल प्रतियोगिता में भाग लेने के लिए प्रोत्साहित करते हैं।
- में भाग लेने CORD-19 Kaggle चैलेंज
- बारे में और जानें COVID -19 ओपन रिसर्च डेटासेट (CORD-19)
- पर दस्तावेज और TF-हब embeddings बारे में अधिक देखें https://tfhub.dev/tensorflow/cord-19/swivel-128d/3
- साथ CORD-19 एम्बेडिंग अंतरिक्ष अन्वेषण TensorFlow एम्बेडिंग प्रोजेक्टर

