 Zobacz na TensorFlow.org Zobacz na TensorFlow.org |  Uruchom w Google Colab Uruchom w Google Colab |  Zobacz na GitHub Zobacz na GitHub |  Pobierz notatnik Pobierz notatnik |  Zobacz model piasty TF Zobacz model piasty TF |
Przewód-19 Obrotowy tekst osadzanie moduł z TF-Hub ( https://tfhub.dev/tensorflow/cord-19/swivel-128d/3 ) został zbudowany w celu wsparcia naukowców analizujących tekst języków naturalnych związanych z COVID-19. Te zanurzeń szkolono na tytuły, autorów, streszczenia, teksty ciała, i tytułów referencyjnych artykułów w CORD-19 zbiorze .
W tej współpracy będziemy:
- Analizuj semantycznie podobne słowa w przestrzeni osadzenia
- Wytrenuj klasyfikatora na zbiorze danych SciCite za pomocą osadzania CORD-19
Ustawiać
import functools
import itertools
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import pandas as pd
import tensorflow as tf
import tensorflow_datasets as tfds
import tensorflow_hub as hub
from tqdm import trange
Przeanalizuj osadzenia
Zacznijmy od analizy osadzania, obliczając i wykreślając macierz korelacji między różnymi terminami. Jeśli osadzanie nauczyło się skutecznie ujmować znaczenie różnych słów, wektory osadzania słów podobnych semantycznie powinny być blisko siebie. Przyjrzyjmy się niektórym terminom związanym z COVID-19.
# Use the inner product between two embedding vectors as the similarity measure
def plot_correlation(labels, features):
corr = np.inner(features, features)
corr /= np.max(corr)
sns.heatmap(corr, xticklabels=labels, yticklabels=labels)
# Generate embeddings for some terms
queries = [
# Related viruses
'coronavirus', 'SARS', 'MERS',
# Regions
'Italy', 'Spain', 'Europe',
# Symptoms
'cough', 'fever', 'throat'
]
module = hub.load('https://tfhub.dev/tensorflow/cord-19/swivel-128d/3')
embeddings = module(queries)
plot_correlation(queries, embeddings)

Widzimy, że osadzanie z powodzeniem uchwyciło znaczenie różnych terminów. Każde słowo jest podobne do innych słów jego klastra (tj. "koronawirus" silnie koreluje z "SARS" i "MERS"), podczas gdy różnią się one od terminów innych klastrów (tj. podobieństwo między "SARS" i "Hiszpania" jest blisko 0).
Zobaczmy teraz, jak możemy wykorzystać te osadzenia do rozwiązania konkretnego zadania.
SciCite: Klasyfikacja intencji cytowania
W tej sekcji pokazano, jak można wykorzystać osadzanie do dalszych zadań, takich jak klasyfikacja tekstu. Użyjemy zestawu danych SciCite z TensorFlow zbiorów danych do sklasyfikowania w intencji cytowań prac naukowych. Mając zdanie z cytatem z pracy naukowej, sklasyfikuj, czy główną intencją cytowania jest podanie informacji ogólnych, zastosowanie metod lub porównanie wyników.
builder = tfds.builder(name='scicite')
builder.download_and_prepare()
train_data, validation_data, test_data = builder.as_dataset(
split=('train', 'validation', 'test'),
as_supervised=True)
Rzućmy okiem na kilka oznaczonych przykładów z zestawu szkoleniowego
NUM_EXAMPLES = 10
TEXT_FEATURE_NAME = builder.info.supervised_keys[0]
LABEL_NAME = builder.info.supervised_keys[1]
def label2str(numeric_label):
m = builder.info.features[LABEL_NAME].names
return m[numeric_label]
data = next(iter(train_data.batch(NUM_EXAMPLES)))
pd.DataFrame({
TEXT_FEATURE_NAME: [ex.numpy().decode('utf8') for ex in data[0]],
LABEL_NAME: [label2str(x) for x in data[1]]
})
Szkolenie klasyfikatora intencji cytatonów
Będziemy trenować klasyfikator na SciCite zestawu danych przy użyciu Keras. Zbudujmy model wykorzystujący embedingi CORD-19 z warstwą klasyfikacyjną na wierzchu.
Hiperparametry
EMBEDDING = 'https://tfhub.dev/tensorflow/cord-19/swivel-128d/3'
TRAINABLE_MODULE = False
hub_layer = hub.KerasLayer(EMBEDDING, input_shape=[],
dtype=tf.string, trainable=TRAINABLE_MODULE)
model = tf.keras.Sequential()
model.add(hub_layer)
model.add(tf.keras.layers.Dense(3))
model.summary()
model.compile(optimizer='adam',
loss=tf.keras.losses.SparseCategoricalCrossentropy(from_logits=True),
metrics=['accuracy'])
Model: "sequential"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
keras_layer (KerasLayer) (None, 128) 17301632
dense (Dense) (None, 3) 387
=================================================================
Total params: 17,302,019
Trainable params: 387
Non-trainable params: 17,301,632
_________________________________________________________________
Trenuj i oceniaj model
Przeszkolmy i oceńmy model, aby zobaczyć wydajność zadania SciCite
EPOCHS = 35
BATCH_SIZE = 32
history = model.fit(train_data.shuffle(10000).batch(BATCH_SIZE),
epochs=EPOCHS,
validation_data=validation_data.batch(BATCH_SIZE),
verbose=1)
Epoch 1/35 257/257 [==============================] - 3s 7ms/step - loss: 0.9244 - accuracy: 0.5924 - val_loss: 0.7915 - val_accuracy: 0.6627 Epoch 2/35 257/257 [==============================] - 2s 5ms/step - loss: 0.7097 - accuracy: 0.7152 - val_loss: 0.6799 - val_accuracy: 0.7358 Epoch 3/35 257/257 [==============================] - 2s 7ms/step - loss: 0.6317 - accuracy: 0.7551 - val_loss: 0.6285 - val_accuracy: 0.7544 Epoch 4/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5938 - accuracy: 0.7687 - val_loss: 0.6032 - val_accuracy: 0.7566 Epoch 5/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5724 - accuracy: 0.7750 - val_loss: 0.5871 - val_accuracy: 0.7653 Epoch 6/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5580 - accuracy: 0.7825 - val_loss: 0.5800 - val_accuracy: 0.7653 Epoch 7/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5484 - accuracy: 0.7870 - val_loss: 0.5711 - val_accuracy: 0.7718 Epoch 8/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5417 - accuracy: 0.7896 - val_loss: 0.5648 - val_accuracy: 0.7806 Epoch 9/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5356 - accuracy: 0.7902 - val_loss: 0.5628 - val_accuracy: 0.7740 Epoch 10/35 257/257 [==============================] - 2s 7ms/step - loss: 0.5313 - accuracy: 0.7903 - val_loss: 0.5581 - val_accuracy: 0.7849 Epoch 11/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5277 - accuracy: 0.7928 - val_loss: 0.5555 - val_accuracy: 0.7838 Epoch 12/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5242 - accuracy: 0.7940 - val_loss: 0.5528 - val_accuracy: 0.7849 Epoch 13/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5215 - accuracy: 0.7947 - val_loss: 0.5522 - val_accuracy: 0.7828 Epoch 14/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5190 - accuracy: 0.7961 - val_loss: 0.5527 - val_accuracy: 0.7751 Epoch 15/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5176 - accuracy: 0.7940 - val_loss: 0.5492 - val_accuracy: 0.7806 Epoch 16/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5154 - accuracy: 0.7978 - val_loss: 0.5500 - val_accuracy: 0.7817 Epoch 17/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5136 - accuracy: 0.7968 - val_loss: 0.5488 - val_accuracy: 0.7795 Epoch 18/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5127 - accuracy: 0.7967 - val_loss: 0.5504 - val_accuracy: 0.7838 Epoch 19/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5111 - accuracy: 0.7970 - val_loss: 0.5470 - val_accuracy: 0.7860 Epoch 20/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5101 - accuracy: 0.7972 - val_loss: 0.5471 - val_accuracy: 0.7871 Epoch 21/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5082 - accuracy: 0.7997 - val_loss: 0.5483 - val_accuracy: 0.7784 Epoch 22/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5077 - accuracy: 0.7995 - val_loss: 0.5471 - val_accuracy: 0.7860 Epoch 23/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5064 - accuracy: 0.8012 - val_loss: 0.5439 - val_accuracy: 0.7871 Epoch 24/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5057 - accuracy: 0.7990 - val_loss: 0.5476 - val_accuracy: 0.7882 Epoch 25/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5050 - accuracy: 0.7996 - val_loss: 0.5442 - val_accuracy: 0.7937 Epoch 26/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5045 - accuracy: 0.7999 - val_loss: 0.5455 - val_accuracy: 0.7860 Epoch 27/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5032 - accuracy: 0.7991 - val_loss: 0.5435 - val_accuracy: 0.7893 Epoch 28/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5034 - accuracy: 0.8022 - val_loss: 0.5431 - val_accuracy: 0.7882 Epoch 29/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5025 - accuracy: 0.8017 - val_loss: 0.5441 - val_accuracy: 0.7937 Epoch 30/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5017 - accuracy: 0.8013 - val_loss: 0.5463 - val_accuracy: 0.7838 Epoch 31/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5015 - accuracy: 0.8017 - val_loss: 0.5453 - val_accuracy: 0.7871 Epoch 32/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5011 - accuracy: 0.8014 - val_loss: 0.5448 - val_accuracy: 0.7915 Epoch 33/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5006 - accuracy: 0.8025 - val_loss: 0.5432 - val_accuracy: 0.7893 Epoch 34/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5005 - accuracy: 0.8008 - val_loss: 0.5448 - val_accuracy: 0.7904 Epoch 35/35 257/257 [==============================] - 2s 5ms/step - loss: 0.4996 - accuracy: 0.8016 - val_loss: 0.5448 - val_accuracy: 0.7915
from matplotlib import pyplot as plt
def display_training_curves(training, validation, title, subplot):
if subplot%10==1: # set up the subplots on the first call
plt.subplots(figsize=(10,10), facecolor='#F0F0F0')
plt.tight_layout()
ax = plt.subplot(subplot)
ax.set_facecolor('#F8F8F8')
ax.plot(training)
ax.plot(validation)
ax.set_title('model '+ title)
ax.set_ylabel(title)
ax.set_xlabel('epoch')
ax.legend(['train', 'valid.'])
display_training_curves(history.history['accuracy'], history.history['val_accuracy'], 'accuracy', 211)
display_training_curves(history.history['loss'], history.history['val_loss'], 'loss', 212)
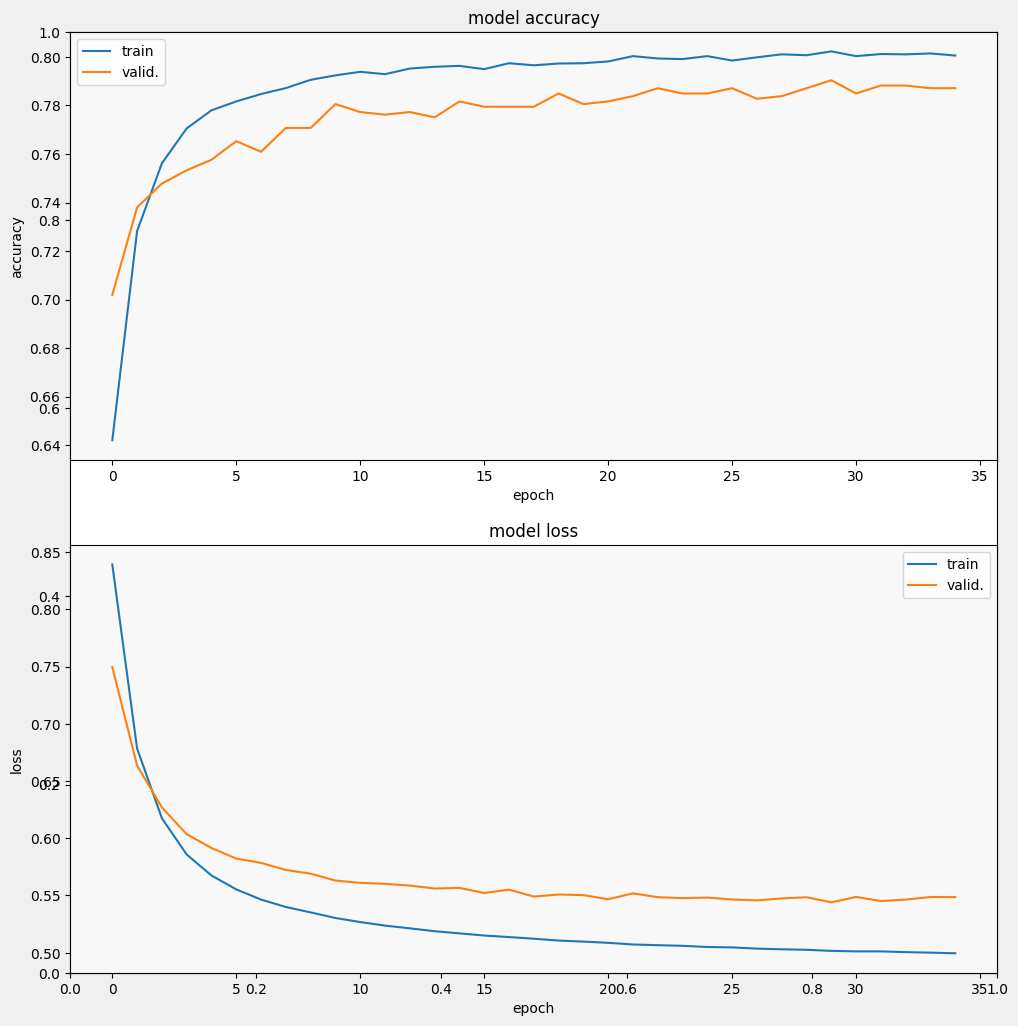
Oceń model
Zobaczmy, jak sprawuje się model. Zostaną zwrócone dwie wartości. Strata (liczba, która reprezentuje nasz błąd, niższe wartości są lepsze) i dokładność.
results = model.evaluate(test_data.batch(512), verbose=2)
for name, value in zip(model.metrics_names, results):
print('%s: %.3f' % (name, value))
4/4 - 0s - loss: 0.5357 - accuracy: 0.7891 - 441ms/epoch - 110ms/step loss: 0.536 accuracy: 0.789
Widzimy, że strata szybko maleje, podczas gdy szczególnie celność szybko rośnie. Narysujmy kilka przykładów, aby sprawdzić, jak prognoza odnosi się do prawdziwych etykiet:
prediction_dataset = next(iter(test_data.batch(20)))
prediction_texts = [ex.numpy().decode('utf8') for ex in prediction_dataset[0]]
prediction_labels = [label2str(x) for x in prediction_dataset[1]]
predictions = [
label2str(x) for x in np.argmax(model.predict(prediction_texts), axis=-1)]
pd.DataFrame({
TEXT_FEATURE_NAME: prediction_texts,
LABEL_NAME: prediction_labels,
'prediction': predictions
})
Widzimy, że dla tej losowej próby model w większości przypadków przewiduje prawidłową etykietę, co wskazuje, że może całkiem dobrze osadzić naukowe zdania.
Co dalej?
Teraz, gdy dowiedziałeś się nieco więcej o osadzaniach CORD-19 Swivel z TF-Hub, zachęcamy Cię do wzięcia udziału w konkursie CORD-19 Kaggle, aby przyczynić się do uzyskania wiedzy naukowej z tekstów akademickich związanych z COVID-19.
- Uczestniczenia w kordu 19 Kaggle wyzwanie
- Dowiedz się więcej o COVID-19 Otwarte Badania Zbiór danych (CORD-19)
- Zobacz dokumentację i więcej o zanurzeń TF-Hub w https://tfhub.dev/tensorflow/cord-19/swivel-128d/3
- Przeglądaj przewód-19 miejsca osadzenia z Osadzanie projektora TensorFlow

