 Voir sur TensorFlow.org Voir sur TensorFlow.org |  Exécuter dans Google Colab Exécuter dans Google Colab |  Voir la source sur GitHub Voir la source sur GitHub |  Télécharger le cahier Télécharger le cahier |
Dans cette collaboration, nous allons explorer l'échantillonnage à partir de la partie postérieure d'un modèle de mélange bayésien gaussien (BGMM) en utilisant uniquement les primitives de probabilité TensorFlow.
Modèle
Pour \(k\in\{1,\ldots, K\}\) composants du mélange chacun de dimension \(D\), nous aimerions modèle \(i\in\{1,\ldots,N\}\) IID échantillons en utilisant le mélange de gaussiennes bayésienne suivant Modèle:
\[\begin{align*} \theta &\sim \text{Dirichlet}(\text{concentration}=\alpha_0)\\ \mu_k &\sim \text{Normal}(\text{loc}=\mu_{0k}, \text{scale}=I_D)\\ T_k &\sim \text{Wishart}(\text{df}=5, \text{scale}=I_D)\\ Z_i &\sim \text{Categorical}(\text{probs}=\theta)\\ Y_i &\sim \text{Normal}(\text{loc}=\mu_{z_i}, \text{scale}=T_{z_i}^{-1/2})\\ \end{align*}\]
Notez les scale arguments ont tous cholesky sémantique. Nous utilisons cette convention car c'est celle de TF Distributions (qui elle-même utilise cette convention en partie parce qu'elle est avantageuse en termes de calcul).
Notre objectif est de générer des échantillons à partir du postérieur :
\[p\left(\theta, \{\mu_k, T_k\}_{k=1}^K \Big| \{y_i\}_{i=1}^N, \alpha_0, \{\mu_{ok}\}_{k=1}^K\right)\]
Notez que \(\{Z_i\}_{i=1}^N\) n'est pas présent - nous sommes des intéressés que les variables aléatoires qui ne redimensionné avec \(N\). (Et heureusement , il y a une distribution de TF qui gère marginalisant sur \(Z_i\).)
Il n'est pas possible d'échantillonner directement à partir de cette distribution en raison d'un terme de normalisation insoluble du point de vue informatique.
Algorithmes Metropolis-Hastings sont pour la technique d'échantillonnage des distributions difficiles à résoudre à Normaliser.
TensorFlow Probability propose un certain nombre d'options MCMC, dont plusieurs basées sur Metropolis-Hastings. Dans ce cahier, nous allons utiliser hamiltonien Monte Carlo ( tfp.mcmc.HamiltonianMonteCarlo ). HMC est souvent un bon choix car il peut converger rapidement, échantillonne l'espace d'état conjointement (par opposition aux coordonnées) et exploite l'une des vertus de TF : la différenciation automatique. Cela dit, à partir d' un échantillonnage postérieur BGMM pourrait effectivement être mieux fait par d' autres approches, par exemple, l'échantillonnage de Gibbs .
%matplotlib inline
import functools
import matplotlib.pyplot as plt; plt.style.use('ggplot')
import numpy as np
import seaborn as sns; sns.set_context('notebook')
import tensorflow.compat.v2 as tf
tf.enable_v2_behavior()
import tensorflow_probability as tfp
tfd = tfp.distributions
tfb = tfp.bijectors
physical_devices = tf.config.experimental.list_physical_devices('GPU')
if len(physical_devices) > 0:
tf.config.experimental.set_memory_growth(physical_devices[0], True)
Avant de construire réellement le modèle, nous devrons définir un nouveau type de distribution. D'après la spécification du modèle ci-dessus, il est clair que nous paramétrons le MVN avec une matrice de covariance inverse, c'est-à-dire [matrice de précision](https://en.wikipedia.org/wiki/Precision_(statistics%29). TF, nous aurons besoin de déployer notre Bijector Ce. Bijector utilisera la transformation avant:
-
Y = tf.linalg.triangular_solve((tf.linalg.matrix_transpose(chol_precision_tril), X, adjoint=True) + loc.
Et le log_prob calcul est juste l'inverse, à savoir:
-
X = tf.linalg.matmul(chol_precision_tril, X - loc, adjoint_a=True).
Étant donné que tous nous avons besoin pour la console HMC est log_prob , cela signifie que nous éviter d' appeler jamais tf.linalg.triangular_solve (comme ce serait le cas pour tfd.MultivariateNormalTriL ). Ceci est avantageux car tf.linalg.matmul est généralement plus rapide due à une meilleure localisation du cache.
class MVNCholPrecisionTriL(tfd.TransformedDistribution):
"""MVN from loc and (Cholesky) precision matrix."""
def __init__(self, loc, chol_precision_tril, name=None):
super(MVNCholPrecisionTriL, self).__init__(
distribution=tfd.Independent(tfd.Normal(tf.zeros_like(loc),
scale=tf.ones_like(loc)),
reinterpreted_batch_ndims=1),
bijector=tfb.Chain([
tfb.Shift(shift=loc),
tfb.Invert(tfb.ScaleMatvecTriL(scale_tril=chol_precision_tril,
adjoint=True)),
]),
name=name)
Le tfd.Independent tours de distribution indépendant tire d'une répartition, en une distribution à plusieurs variables avec des coordonnées statistiquement indépendantes. En termes de calcul log_prob , ce manifeste « méta-distribution » comme une simple somme sur la dimension de l' événement (s).
Notez également que nous avons pris le adjoint ( « Transpose ») de la matrice d'échelle. En effet , si la précision est covariance inverse, c. -à- \(P=C^{-1}\) et si \(C=AA^\top\), puis \(P=BB^{\top}\) où \(B=A^{-\top}\).
Étant donné que cette distribution est un peu difficile, nous allons vérifier rapidement que notre MVNCholPrecisionTriL fonctionne comme nous pensons qu'il devrait.
def compute_sample_stats(d, seed=42, n=int(1e6)):
x = d.sample(n, seed=seed)
sample_mean = tf.reduce_mean(x, axis=0, keepdims=True)
s = x - sample_mean
sample_cov = tf.linalg.matmul(s, s, adjoint_a=True) / tf.cast(n, s.dtype)
sample_scale = tf.linalg.cholesky(sample_cov)
sample_mean = sample_mean[0]
return [
sample_mean,
sample_cov,
sample_scale,
]
dtype = np.float32
true_loc = np.array([1., -1.], dtype=dtype)
true_chol_precision = np.array([[1., 0.],
[2., 8.]],
dtype=dtype)
true_precision = np.matmul(true_chol_precision, true_chol_precision.T)
true_cov = np.linalg.inv(true_precision)
d = MVNCholPrecisionTriL(
loc=true_loc,
chol_precision_tril=true_chol_precision)
[sample_mean, sample_cov, sample_scale] = [
t.numpy() for t in compute_sample_stats(d)]
print('true mean:', true_loc)
print('sample mean:', sample_mean)
print('true cov:\n', true_cov)
print('sample cov:\n', sample_cov)
true mean: [ 1. -1.] sample mean: [ 1.0002806 -1.000105 ] true cov: [[ 1.0625 -0.03125 ] [-0.03125 0.015625]] sample cov: [[ 1.0641273 -0.03126175] [-0.03126175 0.01559312]]
Étant donné que la moyenne et la covariance de l'échantillon sont proches de la vraie moyenne et de la covariance, il semble que la distribution soit correctement implémentée. Maintenant, nous allons utiliser MVNCholPrecisionTriL tfp.distributions.JointDistributionNamed pour spécifier le modèle BGMM. Pour le modèle d' observation, nous allons utiliser tfd.MixtureSameFamily pour intégrer automatiquement le \(\{Z_i\}_{i=1}^N\) dessine.
dtype = np.float64
dims = 2
components = 3
num_samples = 1000
bgmm = tfd.JointDistributionNamed(dict(
mix_probs=tfd.Dirichlet(
concentration=np.ones(components, dtype) / 10.),
loc=tfd.Independent(
tfd.Normal(
loc=np.stack([
-np.ones(dims, dtype),
np.zeros(dims, dtype),
np.ones(dims, dtype),
]),
scale=tf.ones([components, dims], dtype)),
reinterpreted_batch_ndims=2),
precision=tfd.Independent(
tfd.WishartTriL(
df=5,
scale_tril=np.stack([np.eye(dims, dtype=dtype)]*components),
input_output_cholesky=True),
reinterpreted_batch_ndims=1),
s=lambda mix_probs, loc, precision: tfd.Sample(tfd.MixtureSameFamily(
mixture_distribution=tfd.Categorical(probs=mix_probs),
components_distribution=MVNCholPrecisionTriL(
loc=loc,
chol_precision_tril=precision)),
sample_shape=num_samples)
))
def joint_log_prob(observations, mix_probs, loc, chol_precision):
"""BGMM with priors: loc=Normal, precision=Inverse-Wishart, mix=Dirichlet.
Args:
observations: `[n, d]`-shaped `Tensor` representing Bayesian Gaussian
Mixture model draws. Each sample is a length-`d` vector.
mix_probs: `[K]`-shaped `Tensor` representing random draw from
`Dirichlet` prior.
loc: `[K, d]`-shaped `Tensor` representing the location parameter of the
`K` components.
chol_precision: `[K, d, d]`-shaped `Tensor` representing `K` lower
triangular `cholesky(Precision)` matrices, each being sampled from
a Wishart distribution.
Returns:
log_prob: `Tensor` representing joint log-density over all inputs.
"""
return bgmm.log_prob(
mix_probs=mix_probs, loc=loc, precision=chol_precision, s=observations)
Générer des données de « formation »
Pour cette démo, nous allons échantillonner des données aléatoires.
true_loc = np.array([[-2., -2],
[0, 0],
[2, 2]], dtype)
random = np.random.RandomState(seed=43)
true_hidden_component = random.randint(0, components, num_samples)
observations = (true_loc[true_hidden_component] +
random.randn(num_samples, dims).astype(dtype))
Inférence bayésienne à l'aide de HMC
Maintenant que nous avons utilisé TFD pour spécifier notre modèle et obtenu quelques données observées, nous avons tous les éléments nécessaires pour exécuter HMC.
Pour ce faire, nous allons utiliser une application partielle à « Immobilisation » les choses que nous ne voulons pas l' échantillon. Dans ce cas , cela signifie que nous ne ont besoin Immobilisation observations . (L'hyper-paramètres sont déjà cuits dans les distributions a priori et ne fait pas partie de la joint_log_prob signature de fonction).
unnormalized_posterior_log_prob = functools.partial(joint_log_prob, observations)
initial_state = [
tf.fill([components],
value=np.array(1. / components, dtype),
name='mix_probs'),
tf.constant(np.array([[-2., -2],
[0, 0],
[2, 2]], dtype),
name='loc'),
tf.linalg.eye(dims, batch_shape=[components], dtype=dtype, name='chol_precision'),
]
Représentation sans contrainte
L'hamiltonien Monte Carlo (HMC) requiert que la fonction de log-probabilité cible soit différentiable par rapport à ses arguments. De plus, HMC peut présenter une efficacité statistique considérablement plus élevée si l'espace d'état n'est pas contraint.
Cela signifie que nous devrons résoudre deux problèmes principaux lors de l'échantillonnage à partir du postérieur BGMM :
- \(\theta\) représente un vecteur de probabilité discrète, à savoir, doit être telle que \(\sum_{k=1}^K \theta_k = 1\) et \(\theta_k>0\).
- \(T_k\) représente une matrice inverse covariance, ie, doit être telle que \(T_k \succ 0\), à savoir, est définie positive .
Pour répondre à cette exigence, nous devrons :
- transformer les variables contraintes en un espace non contraint
- exécuter le MCMC dans un espace sans contrainte
- transformer les variables non contraintes dans l'espace contraint.
Comme avec MVNCholPrecisionTriL , nous allons utiliser Bijector s pour transformer des variables aléatoires à l' espace sans contrainte.
Le
Dirichletest transformé en un espace sans contrainte via la fonction softmax .Notre variable aléatoire de précision est une distribution sur des matrices semi-définies positives. Pour ceux - ci , nous allons contrainte sur les utiliser
FillTriangularetTransformDiagonalbijectors. Ceux-ci convertissent les vecteurs en matrices triangulaires inférieures et garantissent que la diagonale est positive. Le premier est utile , car elle permet l' échantillonnage ne \(d(d+1)/2\) flotte plutôt que \(d^2\).
unconstraining_bijectors = [
tfb.SoftmaxCentered(),
tfb.Identity(),
tfb.Chain([
tfb.TransformDiagonal(tfb.Softplus()),
tfb.FillTriangular(),
])]
@tf.function(autograph=False)
def sample():
return tfp.mcmc.sample_chain(
num_results=2000,
num_burnin_steps=500,
current_state=initial_state,
kernel=tfp.mcmc.SimpleStepSizeAdaptation(
tfp.mcmc.TransformedTransitionKernel(
inner_kernel=tfp.mcmc.HamiltonianMonteCarlo(
target_log_prob_fn=unnormalized_posterior_log_prob,
step_size=0.065,
num_leapfrog_steps=5),
bijector=unconstraining_bijectors),
num_adaptation_steps=400),
trace_fn=lambda _, pkr: pkr.inner_results.inner_results.is_accepted)
[mix_probs, loc, chol_precision], is_accepted = sample()
Nous allons maintenant exécuter la chaîne et imprimer les moyennes postérieures.
acceptance_rate = tf.reduce_mean(tf.cast(is_accepted, dtype=tf.float32)).numpy()
mean_mix_probs = tf.reduce_mean(mix_probs, axis=0).numpy()
mean_loc = tf.reduce_mean(loc, axis=0).numpy()
mean_chol_precision = tf.reduce_mean(chol_precision, axis=0).numpy()
precision = tf.linalg.matmul(chol_precision, chol_precision, transpose_b=True)
print('acceptance_rate:', acceptance_rate)
print('avg mix probs:', mean_mix_probs)
print('avg loc:\n', mean_loc)
print('avg chol(precision):\n', mean_chol_precision)
acceptance_rate: 0.5305 avg mix probs: [0.25248723 0.60729516 0.1402176 ] avg loc: [[-1.96466753 -2.12047249] [ 0.27628865 0.22944732] [ 2.06461244 2.54216122]] avg chol(precision): [[[ 1.05105032 0. ] [ 0.12699955 1.06553113]] [[ 0.76058015 0. ] [-0.50332767 0.77947431]] [[ 1.22770457 0. ] [ 0.70670027 1.50914164]]]
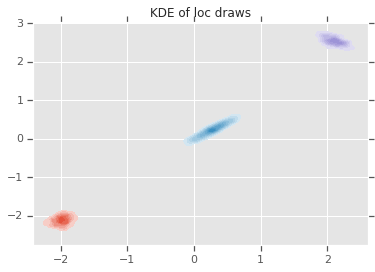
loc_ = loc.numpy()
ax = sns.kdeplot(loc_[:,0,0], loc_[:,0,1], shade=True, shade_lowest=False)
ax = sns.kdeplot(loc_[:,1,0], loc_[:,1,1], shade=True, shade_lowest=False)
ax = sns.kdeplot(loc_[:,2,0], loc_[:,2,1], shade=True, shade_lowest=False)
plt.title('KDE of loc draws');
Conclusion
Cette collaboration simple a montré comment les primitives TensorFlow Probability peuvent être utilisées pour créer des modèles de mélange bayésiens hiérarchiques.
