 Ver en TensorFlow.org Ver en TensorFlow.org |  Ejecutar en Google Colab Ejecutar en Google Colab |  Ver fuente en GitHub Ver fuente en GitHub |  Descargar cuaderno Descargar cuaderno |
TensorFlow Probability (TFP) en JAX ahora tiene herramientas para computación numérica distribuida. Para escalar a un gran número de aceleradores, las herramientas se construyen alrededor de la escritura de código utilizando el paradigma de "datos múltiples de un solo programa", o SPMD para abreviar.
En este cuaderno, veremos cómo "pensar en SPMD" e introduciremos las nuevas abstracciones de TFP para escalar a configuraciones como pods de TPU o clústeres de GPU. Si está ejecutando este código usted mismo, asegúrese de seleccionar un tiempo de ejecución de TPU.
Primero instalaremos las últimas versiones TFP, JAX y TF.
Instala
pip install jaxlib --upgrade -q 2>&1 1> /dev/nullpip install tfp-nightly[jax] --upgrade -q 2>&1 1> /dev/nullpip install tf-nightly-cpu -q -I 2>&1 1> /dev/nullpip install jax -I -q --upgrade 2>&1 1>/dev/null
Importaremos algunas bibliotecas generales, junto con algunas utilidades JAX.
Configuración e importaciones
import functools
import collections
import contextlib
import jax
import jax.numpy as jnp
from jax import lax
from jax import random
import jax.numpy as jnp
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
import pandas as pd
import tensorflow_datasets as tfds
from tensorflow_probability.substrates import jax as tfp
sns.set(style='white')
INFO:tensorflow:Enabling eager execution INFO:tensorflow:Enabling v2 tensorshape INFO:tensorflow:Enabling resource variables INFO:tensorflow:Enabling tensor equality INFO:tensorflow:Enabling control flow v2
También configuraremos algunos alias de TFP útiles. Las nuevas abstracciones se proporcionan actualmente en tfp.experimental.distribute y tfp.experimental.mcmc .
tfd = tfp.distributions
tfb = tfp.bijectors
tfm = tfp.mcmc
tfed = tfp.experimental.distribute
tfde = tfp.experimental.distributions
tfem = tfp.experimental.mcmc
Root = tfed.JointDistributionCoroutine.Root
Para conectar el portátil a una TPU, usamos el siguiente ayudante de JAX. Para confirmar que estamos conectados, imprimimos la cantidad de dispositivos, que debería ser ocho.
from jax.tools import colab_tpu
colab_tpu.setup_tpu()
print(f'Found {jax.device_count()} devices')
Found 8 devices
Una breve introducción a jax.pmap
Después de conectarse a un TPU, tenemos acceso a ocho dispositivos. Sin embargo, cuando ejecutamos código JAX con entusiasmo, JAX por defecto ejecuta cálculos en solo uno.
La forma más sencilla de ejecutar un cálculo en muchos dispositivos es mapear una función, haciendo que cada dispositivo ejecute un índice del mapa. JAX proporciona la jax.pmap ( "mapa paralelo") transformación que convierte una función en uno que se asigna la función de la mayoría de dispositivos.
En el siguiente ejemplo, creamos una matriz de tamaño 8 (para que coincida con la cantidad de dispositivos disponibles) y asignamos una función que agrega 5 a través de ella.
xs = jnp.arange(8.)
out = jax.pmap(lambda x: x + 5.)(xs)
print(type(out), out)
<class 'jax.interpreters.pxla.ShardedDeviceArray'> [ 5. 6. 7. 8. 9. 10. 11. 12.]
Tenga en cuenta que recibimos un ShardedDeviceArray Tipo de nuevo, lo que indica que la matriz de salida se divide físicamente en diferentes dispositivos.
jax.pmap actúa semánticamente como un mapa, pero tiene algunas opciones importantes que modifican su comportamiento. Por defecto, pmap asume todas las entradas de la función se están usando ya, pero podemos modificar este comportamiento con el in_axes argumento.
xs = jnp.arange(8.)
y = 5.
# Map over the 0-axis of `xs` and don't map over `y`
out = jax.pmap(lambda x, y: x + y, in_axes=(0, None))(xs, y)
print(out)
[ 5. 6. 7. 8. 9. 10. 11. 12.]
De forma análoga, la out_axes argumento para pmap determina si volver o no a los valores en todos los dispositivos. Configuración out_axes a None devuelve automáticamente el valor de la primera dispositivo y sólo debe usarse si estamos seguros de los valores son los mismos en todos los dispositivos.
xs = jnp.ones(8) # Value is the same on each device
out = jax.pmap(lambda x: x + 1, out_axes=None)(xs)
print(out)
2.0
¿Qué sucede cuando lo que nos gustaría hacer no se puede expresar fácilmente como una función pura mapeada? Por ejemplo, ¿qué pasa si quisiéramos hacer una suma a lo largo del eje que estamos mapeando? JAX ofrece "colectivos", funciones que se comunican entre dispositivos, para permitir la escritura de programas distribuidos más interesantes y complejos. Para comprender cómo funcionan exactamente, presentaremos SPMD.
¿Qué es SPMD?
Un solo programa de datos múltiples (SPMD) es un modelo de programación concurrente en el que un solo programa (es decir, el mismo código) se ejecuta simultáneamente en todos los dispositivos, pero las entradas a cada uno de los programas en ejecución pueden diferir.
Si nuestro programa es una simple función de sus entradas (es decir, algo así como x + 5 ), se ejecuta un programa en SPMD se acaba de Equivalencia sobre diferentes datos, como lo hicimos con jax.pmap anterior. Sin embargo, podemos hacer más que simplemente "mapear" una función. JAX ofrece "colectivos", que son funciones que se comunican entre dispositivos.
Por ejemplo, tal vez nos gustaría tomar la suma de una cantidad en todos nuestros dispositivos. Antes de hacer eso, tenemos que asignar un nombre a la que se aplica la relación eje sobre el pmap . A continuación, utilizamos el lax.psum función ( "suma paralelo") para realizar una suma en todos los dispositivos, asegurando identificamos el llamado eje que estamos sumando.
def f(x):
out = lax.psum(x, axis_name='i')
return out
xs = jnp.arange(8.) # Length of array matches number of devices
jax.pmap(f, axis_name='i')(xs)
ShardedDeviceArray([28., 28., 28., 28., 28., 28., 28., 28.], dtype=float32)
El psum agregados colectivos el valor de x en cada dispositivo y sincroniza su valor a través del mapa, es decir, out es 28. en cada dispositivo. Ya no estamos realizando un simple "mapa", sino que estamos ejecutando un programa SPMD donde el cálculo de cada dispositivo ahora puede interactuar con el mismo cálculo en otros dispositivos, aunque de forma limitada utilizando colectivos. En este escenario, podemos utilizar out_axes = None , porque psum se sincronizará el valor.
def f(x):
out = lax.psum(x, axis_name='i')
return out
jax.pmap(f, axis_name='i', out_axes=None)(jnp.arange(8.))
ShardedDeviceArray(28., dtype=float32)
SPMD nos permite escribir un programa que se ejecuta en cada dispositivo en cualquier configuración de TPU simultáneamente. El mismo código que se usa para hacer aprendizaje automático en 8 núcleos de TPU se puede usar en un pod de TPU que puede tener cientos o miles de núcleos. Para ver un tutorial más detallada sobre jax.pmap y SPMD, puede hacer referencia a la del JAX 101 tutorial .
MCMC a escala
En este cuaderno, nos centramos en el uso de métodos de Markov Chain Monte Carlo (MCMC) para la inferencia bayesiana. Hay muchas formas en las que utilizamos muchos dispositivos para MCMC, pero en este portátil, nos centraremos en dos:
- Ejecución de cadenas de Markov independientes en diferentes dispositivos. Este caso es bastante simple y se puede hacer con vainilla TFP.
- Fragmentación de un conjunto de datos entre dispositivos. Este caso es un poco más complejo y requiere maquinaria TFP recientemente agregada.
Cadenas independientes
Supongamos que nos gustaría hacer una inferencia bayesiana en un problema utilizando MCMC y nos gustaría ejecutar varias cadenas en paralelo a través de varios dispositivos (digamos 2 en cada dispositivo). Esto resulta ser un programa que simplemente podemos "mapear" entre dispositivos, es decir, uno que no necesita colectivos. Para asegurarnos de que cada programa ejecute una cadena de Markov diferente (en lugar de ejecutar la misma), pasamos un valor diferente para la semilla aleatoria a cada dispositivo.
Probémoslo con un problema de juguete de muestreo a partir de una distribución gaussiana bidimensional. Podemos usar la funcionalidad MCMC existente de TFP lista para usar. En general, intentamos poner la mayor parte de la lógica dentro de nuestra función mapeada para distinguir más explícitamente entre lo que se está ejecutando en todos los dispositivos y solo en el primero.
def run(seed):
target_log_prob = tfd.Sample(tfd.Normal(0., 1.), 2).log_prob
initial_state = jnp.zeros([2, 2]) # 2 chains
kernel = tfm.HamiltonianMonteCarlo(target_log_prob, 1e-1, 10)
def trace_fn(state, pkr):
return target_log_prob(state)
states, log_prob = tfm.sample_chain(
num_results=1000,
num_burnin_steps=1000,
kernel=kernel,
current_state=initial_state,
trace_fn=trace_fn,
seed=seed
)
return states, log_prob
Por sí mismo, el run función toma en una semilla aleatoria sin estado (stateless para ver cómo el trabajo aleatoriedad, se puede leer la PTF en JAX portátil o ver el tutorial JAX 101 ). Mapeo run a través de diferentes semillas resultará en el funcionamiento de varias cadenas de Markov independientes.
states, log_probs = jax.pmap(run)(random.split(random.PRNGKey(0), 8))
print(states.shape, log_probs.shape)
# states is (8 devices, 1000 samples, 2 chains, 2 dimensions)
# log_prob is (8 devices, 1000 samples, 2 chains)
(8, 1000, 2, 2) (8, 1000, 2)
Observe cómo ahora tenemos un eje adicional correspondiente a cada dispositivo. Podemos reorganizar las dimensiones y aplanarlas para obtener un eje para las 16 cadenas.
states = states.transpose([0, 2, 1, 3]).reshape([-1, 1000, 2])
log_probs = log_probs.transpose([0, 2, 1]).reshape([-1, 1000])
fig, ax = plt.subplots(1, 2, figsize=(10, 5))
ax[0].plot(log_probs.T, alpha=0.4)
ax[1].scatter(*states.reshape([-1, 2]).T, alpha=0.1)
plt.show()
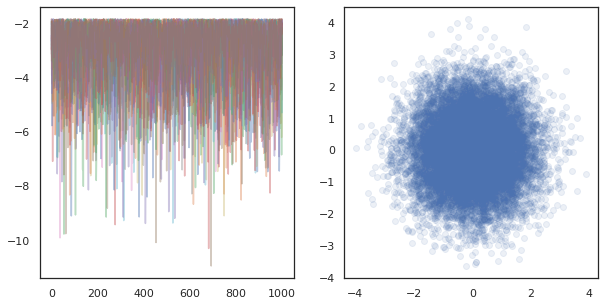
Cuando se ejecuta cadenas independientes en muchos dispositivos, es tan fácil como pmap -ing más de una función que utiliza tfp.mcmc , asegurando que pasan valores diferentes para la semilla aleatoria a cada dispositivo.
Fragmentación de datos
Cuando hacemos MCMC, la distribución objetivo es a menudo una distribución posterior obtenida mediante el condicionamiento de un conjunto de datos, y el cálculo de una densidad logarítmica no normalizada implica la suma de probabilidades para cada dato observado.
Con conjuntos de datos muy grandes, puede resultar prohibitivo incluso ejecutar una cadena en un solo dispositivo. Sin embargo, cuando tenemos acceso a varios dispositivos, podemos dividir el conjunto de datos entre los dispositivos para aprovechar mejor la computación que tenemos disponible.
Si nos gustaría hacer MCMC con un conjunto de datos fragmentada, tenemos que garantizar la no normalizada registro de densidad se calcula en cada dispositivo representa el total, es decir, la densidad sobre todos los datos, de lo contrario cada dispositivo estará haciendo MCMC con su propio destino incorrecto distribución. Con este fin, la PTF tiene ahora nuevas herramientas (es decir tfp.experimental.distribute y tfp.experimental.mcmc ) que permiten utilizar la computadora "fragmentados" probabilidades de registro y hacer MCMC con ellos.
Distribuciones fragmentadas
La abstracción TFP núcleo proporciona ahora para el cálculo de probabiliities de registro fragmentados se la Sharded meta-distribución, que tiene una distribución como entrada y devuelve una nueva distribución que tiene propiedades específicas cuando se ejecuta en un contexto SPMD. Sharded vidas en tfp.experimental.distribute .
Intuitivamente, un Sharded corresponde distribución de un conjunto de variables aleatorias que han sido "dividir" a través de dispositivos. En cada dispositivo, producirán diferentes muestras y pueden tener individualmente diferentes densidades logarítmicas. Alternativamente, un Sharded corresponde de distribución a una "placa" en gráfica modelo de lenguaje, en el que el tamaño de la placa es el número de dispositivos.
Muestreo de un Sharded distribución
Si nos muestra de una Normal de distribución en un ser programa pmap -Ed utilizando la misma semilla en cada dispositivo, obtendremos la misma muestra en cada dispositivo. Podemos pensar en la siguiente función como un muestreo de una única variable aleatoria que está sincronizada entre dispositivos.
# `pmap` expects at least one value to be mapped over, so we provide a dummy one
def f(seed, _):
return tfd.Normal(0., 1.).sample(seed=seed)
jax.pmap(f, in_axes=(None, 0))(random.PRNGKey(0), jnp.arange(8.))
ShardedDeviceArray([-0.20584236, -0.20584236, -0.20584236, -0.20584236,
-0.20584236, -0.20584236, -0.20584236, -0.20584236], dtype=float32)
Si envolvemos tfd.Normal(0., 1.) con un tfed.Sharded , que lógicamente ahora tenemos ocho variables aleatorias diferentes (uno en cada dispositivo) y, por tanto, a producir una muestra diferente para cada uno, a pesar de que pasa en la misma semilla .
def f(seed, _):
return tfed.Sharded(tfd.Normal(0., 1.), shard_axis_name='i').sample(seed=seed)
jax.pmap(f, in_axes=(None, 0), axis_name='i')(random.PRNGKey(0), jnp.arange(8.))
ShardedDeviceArray([ 1.2152631 , 0.7818249 , 0.32549605, 0.6828047 ,
1.3973192 , -0.57830244, 0.37862757, 2.7706041 ], dtype=float32)
Una representación equivalente de esta distribución en un solo dispositivo son solo 8 muestras normales independientes. A pesar de que el valor de la muestra será diferente ( tfed.Sharded hace pseudo-aleatoria de generación de números de forma ligeramente diferente), que ambos representan la misma distribución.
dist = tfd.Sample(tfd.Normal(0., 1.), jax.device_count())
dist.sample(seed=random.PRNGKey(0))
DeviceArray([ 0.08086783, -0.38624594, -0.3756545 , 1.668957 ,
-1.2758069 , 2.1192007 , -0.85821325, 1.1305912 ], dtype=float32)
Tomando el registro de densidad de un Sharded distribución
Veamos qué sucede cuando calculamos la densidad logarítmica de una muestra a partir de una distribución regular en un contexto SPMD.
def f(seed, _):
dist = tfd.Normal(0., 1.)
x = dist.sample(seed=seed)
return x, dist.log_prob(x)
jax.pmap(f, in_axes=(None, 0))(random.PRNGKey(0), jnp.arange(8.))
(ShardedDeviceArray([-0.20584236, -0.20584236, -0.20584236, -0.20584236,
-0.20584236, -0.20584236, -0.20584236, -0.20584236], dtype=float32),
ShardedDeviceArray([-0.94012403, -0.94012403, -0.94012403, -0.94012403,
-0.94012403, -0.94012403, -0.94012403, -0.94012403], dtype=float32))
Cada muestra es la misma en cada dispositivo, por lo que también calculamos la misma densidad en cada dispositivo. Intuitivamente, aquí solo tenemos una distribución sobre una única variable distribuida normalmente.
Con un Sharded distribución, tenemos una distribución de más de 8 variables aleatorias, así que cuando se calcula la log_prob de una muestra, sumamos, a través de dispositivos, sobre cada una de las densidades de registro individuales. (Puede notar que este valor total de log_prob es mayor que el singleton log_prob calculado anteriormente).
def f(seed, _):
dist = tfed.Sharded(tfd.Normal(0., 1.), shard_axis_name='i')
x = dist.sample(seed=seed)
return x, dist.log_prob(x)
sample, log_prob = jax.pmap(f, in_axes=(None, 0), axis_name='i')(
random.PRNGKey(0), jnp.arange(8.))
print('Sample:', sample)
print('Log Prob:', log_prob)
Sample: [ 1.2152631 0.7818249 0.32549605 0.6828047 1.3973192 -0.57830244 0.37862757 2.7706041 ] Log Prob: [-13.7349205 -13.7349205 -13.7349205 -13.7349205 -13.7349205 -13.7349205 -13.7349205 -13.7349205]
La distribución equivalente "sin fragmentar" produce la misma densidad logarítmica.
dist = tfd.Sample(tfd.Normal(0., 1.), jax.device_count())
dist.log_prob(sample)
DeviceArray(-13.7349205, dtype=float32)
A Sharded distribución produce valores diferentes de sample en cada dispositivo, pero conseguir el mismo valor para log_prob en cada dispositivo. ¿Que esta pasando aqui? A Sharded distribución hace un psum internamente para asegurar los log_prob valores están sincronizados a través de dispositivos. ¿Por qué querríamos este comportamiento? Si nos estamos quedando la misma cadena MCMC en cada dispositivo, nos gustaría que la target_log_prob a ser la misma en cada dispositivo, incluso si algunas variables aleatorias en el cálculo están fragmentados en todos los dispositivos.
Además, un Sharded garantiza una distribución que los gradientes a través de dispositivos son la correcta, para asegurar que los algoritmos como HMC, que tienen gradientes de la función de registro de densidad como parte de la función de transición, producen muestras adecuadas.
Fragmentada JointDistribution s
Podemos crear modelos con múltiples Sharded variables aleatorias mediante el uso de JointDistribution s (JDs). Por desgracia, Sharded distribuciones no se pueden utilizar de manera segura con la vainilla tfd.JointDistribution s, pero tfp.experimental.distribute exportaciones "parches" JDs que se comportará como Sharded distribuciones.
def f(seed, _):
dist = tfed.JointDistributionSequential([
tfd.Normal(0., 1.),
tfed.Sharded(tfd.Normal(0., 1.), shard_axis_name='i'),
])
x = dist.sample(seed=seed)
return x, dist.log_prob(x)
jax.pmap(f, in_axes=(None, 0), axis_name='i')(random.PRNGKey(0), jnp.arange(8.))
([ShardedDeviceArray([1.6121525, 1.6121525, 1.6121525, 1.6121525, 1.6121525,
1.6121525, 1.6121525, 1.6121525], dtype=float32),
ShardedDeviceArray([ 0.8690128 , -0.83167845, 1.2209264 , 0.88412696,
0.76478404, -0.66208494, -0.0129658 , 0.7391483 ], dtype=float32)],
ShardedDeviceArray([-12.214451, -12.214451, -12.214451, -12.214451,
-12.214451, -12.214451, -12.214451, -12.214451], dtype=float32))
Estos JDs fragmentados pueden tener tanto Sharded distribuciones y vainilla PTF como componentes. Para las distribuciones no fragmentadas, obtenemos la misma muestra en cada dispositivo, y para las distribuciones fragmentadas, obtenemos muestras diferentes. El log_prob en cada dispositivo se sincroniza también.
MCMC con Sharded distribuciones
¿Cómo podemos pensar en Sharded distribuciones en el contexto de MCMC? Si tenemos un modelo generativo que puede ser expresado como una JointDistribution , podemos recoger algunas eje de ese modelo de "fragmento" de diámetro. Por lo general, una variable aleatoria en el modelo corresponderá a los datos observados, y si tenemos un conjunto de datos grande que nos gustaría dividir entre dispositivos, queremos que las variables asociadas a los puntos de datos también se fragmenten. También podemos tener variables aleatorias "locales" que son uno a uno con las observaciones que estamos fragmentando, por lo que tendremos que fragmentar adicionalmente esas variables aleatorias.
Vamos a repasar ejemplos del uso de la Sharded distribuciones con la PTF MCMC en esta sección. Vamos a empezar con un ejemplo de regresión logística bayesiana simple, y concluimos con un ejemplo factorización de la matriz, con el objetivo de demostrar algunos casos de uso para el distribute biblioteca.
Ejemplo: regresión logística bayesiana para MNIST
Nos gustaría hacer una regresión logística bayesiana en un gran conjunto de datos; el modelo tiene una antes \(p(\theta)\) largo de los pesos de regresión, y una probabilidad \(p(y_i | \theta, x_i)\) que se suma sobre todos los datos \(\{x_i, y_i\}_{i = 1}^N\) para obtener la densidad total de registro de articulación. Si nos fragmento nuestros datos, nos Shard las variables aleatorias observadas \(x_i\) y \(y_i\) en nuestro modelo.
Usamos el siguiente modelo de regresión logística bayesiana para la clasificación MNIST:
\[ \begin{align*} w &\sim \mathcal{N}(0, 1) \\ b &\sim \mathcal{N}(0, 1) \\ y_i | w, b, x_i &\sim \textrm{Categorical}(w^T x_i + b) \end{align*} \]
Carguemos MNIST usando conjuntos de datos de TensorFlow.
mnist = tfds.as_numpy(tfds.load('mnist', batch_size=-1))
raw_train_images, train_labels = mnist['train']['image'], mnist['train']['label']
train_images = raw_train_images.reshape([raw_train_images.shape[0], -1]) / 255.
raw_test_images, test_labels = mnist['test']['image'], mnist['test']['label']
test_images = raw_test_images.reshape([raw_test_images.shape[0], -1]) / 255.
Downloading and preparing dataset mnist/3.0.1 (download: 11.06 MiB, generated: 21.00 MiB, total: 32.06 MiB) to /root/tensorflow_datasets/mnist/3.0.1... WARNING:absl:Dataset mnist is hosted on GCS. It will automatically be downloaded to your local data directory. If you'd instead prefer to read directly from our public GCS bucket (recommended if you're running on GCP), you can instead pass `try_gcs=True` to `tfds.load` or set `data_dir=gs://tfds-data/datasets`. HBox(children=(FloatProgress(value=0.0, description='Dl Completed...', max=4.0, style=ProgressStyle(descriptio… Dataset mnist downloaded and prepared to /root/tensorflow_datasets/mnist/3.0.1. Subsequent calls will reuse this data.
Tenemos 60000 imágenes de entrenamiento, pero aprovechemos nuestros 8 núcleos disponibles y dividámoslo en 8 formas. Vamos a utilizar este práctico shard función de utilidad.
def shard_value(x):
x = x.reshape((jax.device_count(), -1, *x.shape[1:]))
return jax.pmap(lambda x: x)(x) # pmap will physically place values on devices
shard = functools.partial(jax.tree_map, shard_value)
sharded_train_images, sharded_train_labels = shard((train_images, train_labels))
print(sharded_train_images.shape, sharded_train_labels.shape)
(8, 7500, 784) (8, 7500)
Antes de continuar, analicemos rápidamente la precisión en las TPU y su impacto en la HMC. TPU ejecutar multiplicaciones de matrices utilizando baja bfloat16 de precisión para la velocidad. bfloat16 multiplicaciones de matrices son a menudo suficientes para muchas aplicaciones de aprendizaje profundos, pero cuando se utiliza con HMC, se han encontrado empíricamente la precisión inferior puede conducir a trayectorias divergentes, provocando rechazos. Podemos utilizar multiplicaciones de matrices de mayor precisión, a costa de algunos cálculos adicionales.
Para aumentar nuestra precisión matmul, podemos utilizar el jax.default_matmul_precision decorador con "tensorfloat32" de precisión (para aumentar aún más la precisión que podríamos utilizar "float32" precisión).
Ahora vamos a definir nuestra run función, que se llevará en una semilla aleatoria (que será el mismo en cada dispositivo) y un fragmento de MNIST. La función implementará el modelo mencionado anteriormente y luego usaremos la funcionalidad básica MCMC de TFP para ejecutar una sola cadena. Nos aseguraremos de que para decorar run con el jax.default_matmul_precision decorador para asegurarse de que la multiplicación de matrices se ejecuta con mayor precisión, aunque en el ejemplo concreto continuación, sólo así podríamos utilizar jnp.dot(images, w, precision=lax.Precision.HIGH) .
# We can use `out_axes=None` in the `pmap` because the results will be the same
# on every device.
@functools.partial(jax.pmap, axis_name='data', in_axes=(None, 0), out_axes=None)
@jax.default_matmul_precision('tensorfloat32')
def run(seed, data):
images, labels = data # a sharded dataset
num_examples, dim = images.shape
num_classes = 10
def model_fn():
w = yield Root(tfd.Sample(tfd.Normal(0., 1.), [dim, num_classes]))
b = yield Root(tfd.Sample(tfd.Normal(0., 1.), [num_classes]))
logits = jnp.dot(images, w) + b
yield tfed.Sharded(tfd.Independent(tfd.Categorical(logits=logits), 1),
shard_axis_name='data')
model = tfed.JointDistributionCoroutine(model_fn)
init_seed, sample_seed = random.split(seed)
initial_state = model.sample(seed=init_seed)[:-1] # throw away `y`
def target_log_prob(*state):
return model.log_prob((*state, labels))
def accuracy(w, b):
logits = images.dot(w) + b
preds = logits.argmax(axis=-1)
# We take the average accuracy across devices by using `lax.pmean`
return lax.pmean((preds == labels).mean(), 'data')
kernel = tfm.HamiltonianMonteCarlo(target_log_prob, 1e-2, 100)
kernel = tfm.DualAveragingStepSizeAdaptation(kernel, 500)
def trace_fn(state, pkr):
return (
target_log_prob(*state),
accuracy(*state),
pkr.new_step_size)
states, trace = tfm.sample_chain(
num_results=1000,
num_burnin_steps=1000,
current_state=initial_state,
kernel=kernel,
trace_fn=trace_fn,
seed=sample_seed
)
return states, trace
jax.pmap incluye una compilación JIT pero la función compilado se almacena en caché después de la primera llamada. Llamaremos a run e ignoramos la salida para almacenar en caché la compilación.
%%time
output = run(random.PRNGKey(0), (sharded_train_images, sharded_train_labels))
jax.tree_map(lambda x: x.block_until_ready(), output)
CPU times: user 24.5 s, sys: 48.2 s, total: 1min 12s Wall time: 1min 54s
Ahora llamaré a run de nuevo para ver cuánto tiempo tarda la ejecución real.
%%time
states, trace = run(random.PRNGKey(0), (sharded_train_images, sharded_train_labels))
jax.tree_map(lambda x: x.block_until_ready(), trace)
CPU times: user 13.1 s, sys: 45.2 s, total: 58.3 s Wall time: 1min 43s
Estamos ejecutando 200.000 pasos de salto, cada uno de los cuales calcula un gradiente en todo el conjunto de datos. Dividir el cálculo en 8 núcleos nos permite calcular el equivalente a 200.000 épocas de entrenamiento en unos 95 segundos, ¡unas 2.100 épocas por segundo!
Grafiquemos la densidad logarítmica de cada muestra y la precisión de cada muestra:
fig, ax = plt.subplots(1, 3, figsize=(15, 5))
ax[0].plot(trace[0])
ax[0].set_title('Log Prob')
ax[1].plot(trace[1])
ax[1].set_title('Accuracy')
ax[2].plot(trace[2])
ax[2].set_title('Step Size')
plt.show()
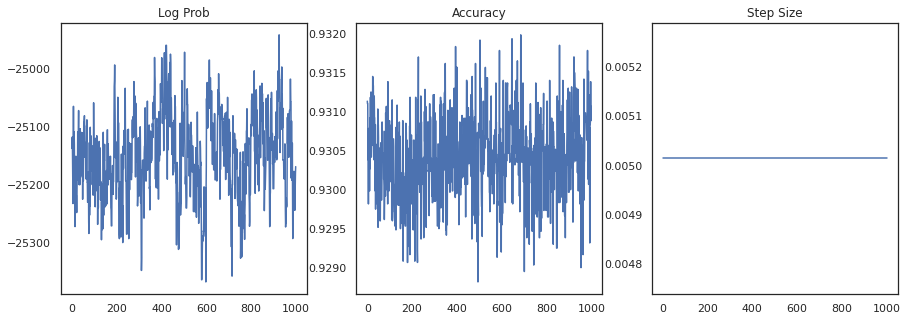
Si ensamblamos las muestras, podemos calcular un promedio del modelo bayesiano para mejorar nuestro desempeño.
@functools.partial(jax.pmap, axis_name='data', in_axes=(0, None), out_axes=None)
def bayesian_model_average(data, states):
images, labels = data
logits = jax.vmap(lambda w, b: images.dot(w) + b)(*states)
probs = jax.nn.softmax(logits, axis=-1)
bma_accuracy = (probs.mean(axis=0).argmax(axis=-1) == labels).mean()
avg_accuracy = (probs.argmax(axis=-1) == labels).mean()
return lax.pmean(bma_accuracy, axis_name='data'), lax.pmean(avg_accuracy, axis_name='data')
sharded_test_images, sharded_test_labels = shard((test_images, test_labels))
bma_acc, avg_acc = bayesian_model_average((sharded_test_images, sharded_test_labels), states)
print(f'Average Accuracy: {avg_acc}')
print(f'BMA Accuracy: {bma_acc}')
print(f'Accuracy Improvement: {bma_acc - avg_acc}')
Average Accuracy: 0.9188529253005981 BMA Accuracy: 0.9264000058174133 Accuracy Improvement: 0.0075470805168151855
¡El promedio de un modelo bayesiano aumenta nuestra precisión en casi un 1%!
Ejemplo: sistema de recomendación MovieLens
Intentemos ahora hacer inferencias con el conjunto de datos de recomendaciones de MovieLens, que es una colección de usuarios y sus calificaciones de varias películas. Específicamente, podemos representar MovieLens como un \(N \times M\) matriz reloj \(W\) donde \(N\) es el número de usuarios y \(M\) es el número de películas; esperamos \(N > M\). Las entradas de \(W_{ij}\) son de un valor booleano que indica si o no el usuario \(i\) observaban película \(j\). Tenga en cuenta que MovieLens proporciona calificaciones de los usuarios, pero las ignoramos para simplificar el problema.
Primero, cargaremos el conjunto de datos. Usaremos la versión con 1 millón de calificaciones.
movielens = tfds.as_numpy(tfds.load('movielens/1m-ratings', batch_size=-1))
GENRES = ['Action', 'Adventure', 'Animation', 'Children', 'Comedy',
'Crime', 'Documentary', 'Drama', 'Fantasy', 'Film-Noir',
'Horror', 'IMAX', 'Musical', 'Mystery', 'Romance', 'Sci-Fi',
'Thriller', 'Unknown', 'War', 'Western', '(no genres listed)']
Downloading and preparing dataset movielens/1m-ratings/0.1.0 (download: Unknown size, generated: Unknown size, total: Unknown size) to /root/tensorflow_datasets/movielens/1m-ratings/0.1.0... HBox(children=(FloatProgress(value=1.0, bar_style='info', description='Dl Completed...', max=1.0, style=Progre… HBox(children=(FloatProgress(value=1.0, bar_style='info', description='Dl Size...', max=1.0, style=ProgressSty… HBox(children=(FloatProgress(value=1.0, bar_style='info', description='Extraction completed...', max=1.0, styl… HBox(children=(FloatProgress(value=1.0, bar_style='info', max=1.0), HTML(value=''))) Shuffling and writing examples to /root/tensorflow_datasets/movielens/1m-ratings/0.1.0.incompleteYKA3TG/movielens-train.tfrecord HBox(children=(FloatProgress(value=0.0, max=1000209.0), HTML(value=''))) Dataset movielens downloaded and prepared to /root/tensorflow_datasets/movielens/1m-ratings/0.1.0. Subsequent calls will reuse this data.
Haremos un poco de pre-procesamiento de la base de datos para obtener la matriz reloj \(W\).
raw_movie_ids = movielens['train']['movie_id']
raw_user_ids = movielens['train']['user_id']
genres = movielens['train']['movie_genres']
movie_ids, movie_labels = pd.factorize(movielens['train']['movie_id'])
user_ids, user_labels = pd.factorize(movielens['train']['user_id'])
num_movies = movie_ids.max() + 1
num_users = user_ids.max() + 1
movie_titles = dict(zip(movielens['train']['movie_id'],
movielens['train']['movie_title']))
movie_genres = dict(zip(movielens['train']['movie_id'],
genres))
movie_id_to_title = [movie_titles[movie_labels[id]].decode('utf-8')
for id in range(num_movies)]
movie_id_to_genre = [GENRES[movie_genres[movie_labels[id]][0]] for id in range(num_movies)]
watch_matrix = np.zeros((num_users, num_movies), bool)
watch_matrix[user_ids, movie_ids] = True
print(watch_matrix.shape)
(6040, 3706)
Podemos definir un modelo generativo para \(W\), usando un modelo simple de la factorización de la matriz probabilística. Se parte de una latente \(N \times D\) matriz usuario \(U\) y una latente \(M \times D\) matriz de película \(V\), que cuando se multiplica producir los logits de Bernoulli para la matriz reloj \(W\). También incluiremos unos vectores de polarización para los usuarios y películas, \(u\) y \(v\).
\[ \begin{align*} U &\sim \mathcal{N}(0, 1) \quad u \sim \mathcal{N}(0, 1)\\ V &\sim \mathcal{N}(0, 1) \quad v \sim \mathcal{N}(0, 1)\\ W_{ij} &\sim \textrm{Bernoulli}\left(\sigma\left(\left(UV^T\right)_{ij} + u_i + v_j\right)\right) \end{align*} \]
Esta es una matriz bastante grande; 6040 usuarios y 3706 películas conducen a una matriz con más de 22 millones de entradas. ¿Cómo abordamos la fragmentación de este modelo? Pues bien, si asumimos que \(N > M\) (es decir, hay más usuarios que las películas), entonces tendría sentido para fragmentar la matriz reloj a través del eje de usuario, por lo que cada dispositivo tendría un trozo de matriz de reloj correspondiente a un subconjunto de usuarios . A diferencia del ejemplo anterior, sin embargo, que también tendrá que fragmentar el \(U\) matriz, ya que tiene una incrustación para cada usuario, por lo que cada dispositivo será responsable de un fragmento de \(U\) y un fragmento de \(W\). Por otro lado, \(V\) será unsharded y ser sincronizado a través de dispositivos.
sharded_watch_matrix = shard(watch_matrix)
Antes de escribir nuestra run , vamos a discutir con rapidez los retos adicionales con sharding la variable aleatoria locales \(U\). Cuando se ejecuta HMC, la vainilla tfp.mcmc.HamiltonianMonteCarlo kernel tomará muestras de movimiento para cada elemento del estado de la cadena. Anteriormente, solo las variables aleatorias no fragmentadas formaban parte de ese estado, y los momentos eran los mismos en cada dispositivo. Cuando ahora tenemos una fragmentada \(U\), tenemos que probar diferentes momentos en cada dispositivo para \(U\), mientras que el muestreo de la misma ímpetus para \(V\). Para lograr esto, podemos utilizar tfp.experimental.mcmc.PreconditionedHamiltonianMonteCarlo con una Sharded distribución de los impulsos. A medida que continuamos haciendo el cálculo paralelo de primera clase, podemos simplificar esto, por ejemplo, llevando un indicador de fragmentación al kernel de HMC.
def make_run(*,
axis_name,
dim=20,
num_chains=2,
prior_variance=1.,
step_size=1e-2,
num_leapfrog_steps=100,
num_burnin_steps=1000,
num_results=500,
):
@functools.partial(jax.pmap, in_axes=(None, 0), axis_name=axis_name)
@jax.default_matmul_precision('tensorfloat32')
def run(key, watch_matrix):
num_users, num_movies = watch_matrix.shape
Sharded = functools.partial(tfed.Sharded, shard_axis_name=axis_name)
def prior_fn():
user_embeddings = yield Root(Sharded(tfd.Sample(tfd.Normal(0., 1.), [num_users, dim]), name='user_embeddings'))
user_bias = yield Root(Sharded(tfd.Sample(tfd.Normal(0., 1.), [num_users]), name='user_bias'))
movie_embeddings = yield Root(tfd.Sample(tfd.Normal(0., 1.), [num_movies, dim], name='movie_embeddings'))
movie_bias = yield Root(tfd.Sample(tfd.Normal(0., 1.), [num_movies], name='movie_bias'))
return (user_embeddings, user_bias, movie_embeddings, movie_bias)
prior = tfed.JointDistributionCoroutine(prior_fn)
def model_fn():
user_embeddings, user_bias, movie_embeddings, movie_bias = yield from prior_fn()
logits = (jnp.einsum('...nd,...md->...nm', user_embeddings, movie_embeddings)
+ user_bias[..., :, None] + movie_bias[..., None, :])
yield Sharded(tfd.Independent(tfd.Bernoulli(logits=logits), 2), name='watch')
model = tfed.JointDistributionCoroutine(model_fn)
init_key, sample_key = random.split(key)
initial_state = prior.sample(seed=init_key, sample_shape=num_chains)
def target_log_prob(*state):
return model.log_prob((*state, watch_matrix))
momentum_distribution = tfed.JointDistributionSequential([
Sharded(tfd.Independent(tfd.Normal(jnp.zeros([num_chains, num_users, dim]), 1.), 2)),
Sharded(tfd.Independent(tfd.Normal(jnp.zeros([num_chains, num_users]), 1.), 1)),
tfd.Independent(tfd.Normal(jnp.zeros([num_chains, num_movies, dim]), 1.), 2),
tfd.Independent(tfd.Normal(jnp.zeros([num_chains, num_movies]), 1.), 1),
])
# We pass in momentum_distribution here to ensure that the momenta for
# user_embeddings and user_bias are also sharded
kernel = tfem.PreconditionedHamiltonianMonteCarlo(target_log_prob, step_size,
num_leapfrog_steps,
momentum_distribution=momentum_distribution)
num_adaptation_steps = int(0.8 * num_burnin_steps)
kernel = tfm.DualAveragingStepSizeAdaptation(kernel, num_adaptation_steps)
def trace_fn(state, pkr):
return {
'log_prob': target_log_prob(*state),
'log_accept_ratio': pkr.inner_results.log_accept_ratio,
}
return tfm.sample_chain(
num_results, initial_state,
kernel=kernel,
num_burnin_steps=num_burnin_steps,
trace_fn=trace_fn,
seed=sample_key)
return run
Vamos a correr de nuevo una vez para almacenar en caché el compilado run .
%%time
run = make_run(axis_name='data')
output = run(random.PRNGKey(0), sharded_watch_matrix)
jax.tree_map(lambda x: x.block_until_ready(), output)
CPU times: user 56 s, sys: 1min 24s, total: 2min 20s Wall time: 3min 35s
Ahora lo ejecutaremos de nuevo sin la sobrecarga de compilación.
%%time
states, trace = run(random.PRNGKey(0), sharded_watch_matrix)
jax.tree_map(lambda x: x.block_until_ready(), trace)
CPU times: user 28.8 s, sys: 1min 16s, total: 1min 44s Wall time: 3min 1s
Parece que completamos unos 150.000 pasos de salto en unos 3 minutos, ¡unos 83 pasos de salto por segundo! Grafiquemos la relación de aceptación y la densidad logarítmica de nuestras muestras.
fig, axs = plt.subplots(1, len(trace), figsize=(5 * len(trace), 5))
for ax, (key, val) in zip(axs, trace.items()):
ax.plot(val[0]) # Indexing into a sharded array, each element is the same
ax.set_title(key);
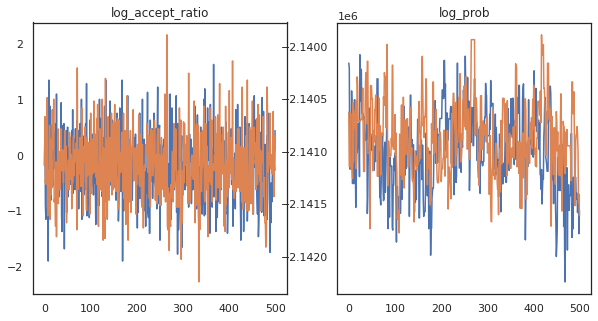
Ahora que tenemos algunas muestras de nuestra cadena de Markov, usémoslas para hacer algunas predicciones. Primero, extraigamos cada uno de los componentes. Recuerde que los user_embeddings y user_bias son división a través de dispositivo, por lo que necesitamos para concatenar nuestra ShardedArray para obtener todos ellos. Por otro lado, movie_embeddings y movie_bias son los mismos en todos los dispositivos, por lo que sólo puede recoger el valor del primer fragmento. Vamos a utilizar regularmente numpy para copiar los valores de la parte posterior TPU de la CPU.
user_embeddings = np.concatenate(np.array(states.user_embeddings, np.float32), axis=2)
user_bias = np.concatenate(np.array(states.user_bias, np.float32), axis=2)
movie_embeddings = np.array(states.movie_embeddings[0], dtype=np.float32)
movie_bias = np.array(states.movie_bias[0], dtype=np.float32)
samples = (user_embeddings, user_bias, movie_embeddings, movie_bias)
print(f'User embeddings: {user_embeddings.shape}')
print(f'User bias: {user_bias.shape}')
print(f'Movie embeddings: {movie_embeddings.shape}')
print(f'Movie bias: {movie_bias.shape}')
User embeddings: (500, 2, 6040, 20) User bias: (500, 2, 6040) Movie embeddings: (500, 2, 3706, 20) Movie bias: (500, 2, 3706)
Intentemos construir un sistema de recomendación simple que utilice la incertidumbre capturada en estas muestras. Primero escribamos una función que clasifique las películas según la probabilidad de visualización.
@jax.jit
def recommend(sample, user_id):
user_embeddings, user_bias, movie_embeddings, movie_bias = sample
movie_logits = (
jnp.einsum('d,md->m', user_embeddings[user_id], movie_embeddings)
+ user_bias[user_id] + movie_bias)
return movie_logits.argsort()[::-1]
Ahora podemos escribir una función que recorra todas las muestras y, para cada una, elija la película mejor clasificada que el usuario aún no haya visto. Luego, podemos ver el recuento de todas las películas recomendadas en las muestras.
def get_recommendations(user_id):
movie_ids = []
already_watched = set(jnp.arange(num_movies)[watch_matrix[user_id] == 1])
for i in range(500):
for j in range(2):
sample = jax.tree_map(lambda x: x[i, j], samples)
ranking = recommend(sample, user_id)
for movie_id in ranking:
if int(movie_id) not in already_watched:
movie_ids.append(movie_id)
break
return movie_ids
def plot_recommendations(movie_ids, ax=None):
titles = collections.Counter([movie_id_to_title[i] for i in movie_ids])
ax = ax or plt.gca()
names, counts = zip(*sorted(titles.items(), key=lambda x: -x[1]))
ax.bar(names, counts)
ax.set_xticklabels(names, rotation=90)
Consideremos al usuario que ha visto más películas frente al que ha visto menos.
user_watch_counts = watch_matrix.sum(axis=1)
user_most = user_watch_counts.argmax()
user_least = user_watch_counts.argmin()
print(user_watch_counts[user_most], user_watch_counts[user_least])
2314 20
Esperamos que nuestro sistema tiene más certeza sobre user_most que user_least , dado que tenemos más información sobre qué tipo de películas user_most es más probable que mirar.
fig, ax = plt.subplots(1, 2, figsize=(20, 10))
most_recommendations = get_recommendations(user_most)
plot_recommendations(most_recommendations, ax=ax[0])
ax[0].set_title('Recommendation for user_most')
least_recommendations = get_recommendations(user_least)
plot_recommendations(least_recommendations, ax=ax[1])
ax[1].set_title('Recommendation for user_least');
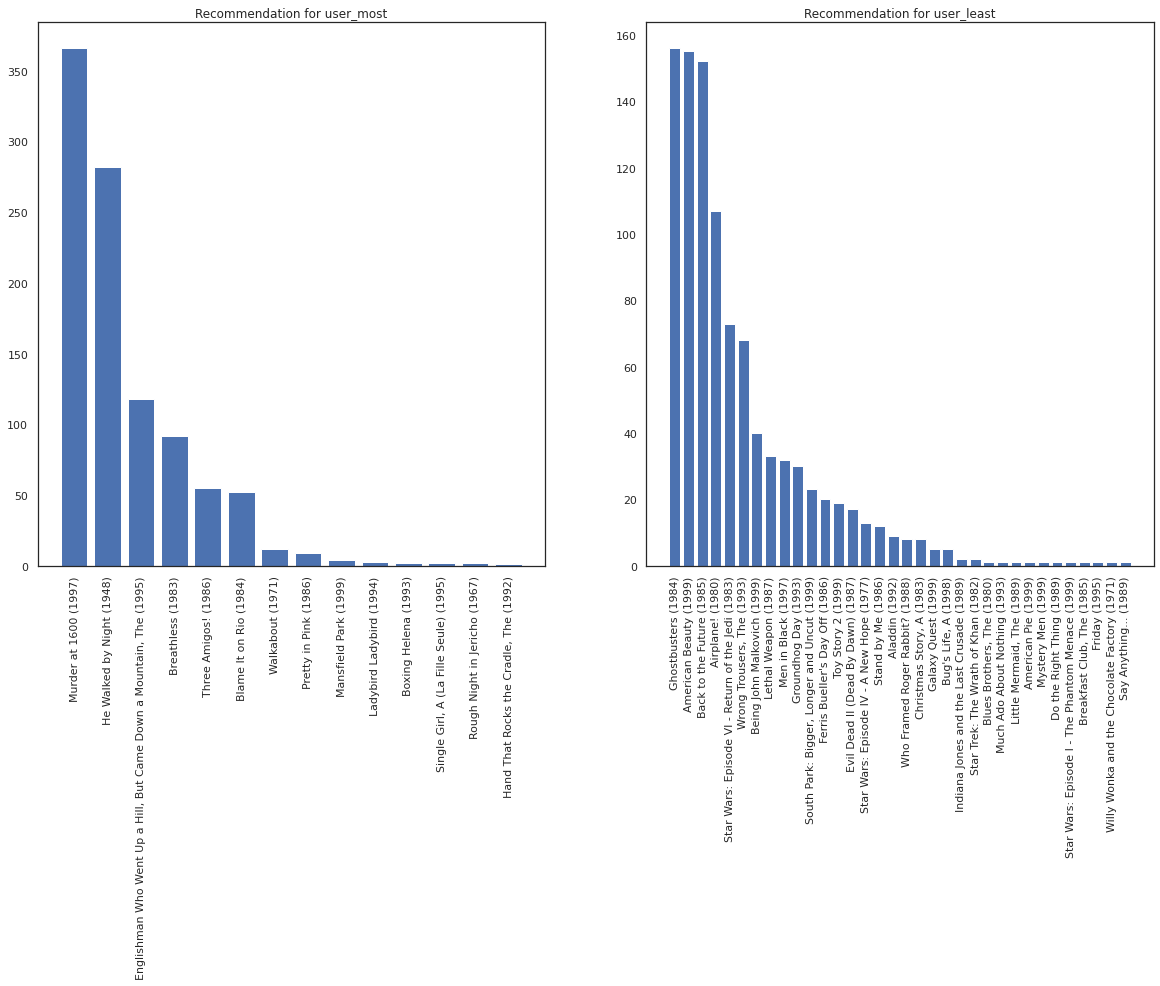
Vemos que hay más variación en nuestras recomendaciones para user_least que refleja nuestra incertidumbre adicional en sus preferencias de vigilancia.
También podemos ver mirar los géneros de las películas recomendadas.
most_genres = collections.Counter([movie_id_to_genre[i] for i in most_recommendations])
least_genres = collections.Counter([movie_id_to_genre[i] for i in least_recommendations])
fig, ax = plt.subplots(1, 2, figsize=(20, 10))
ax[0].bar(most_genres.keys(), most_genres.values())
ax[0].set_title('Genres recommended for user_most')
ax[1].bar(least_genres.keys(), least_genres.values())
ax[1].set_title('Genres recommended for user_least');
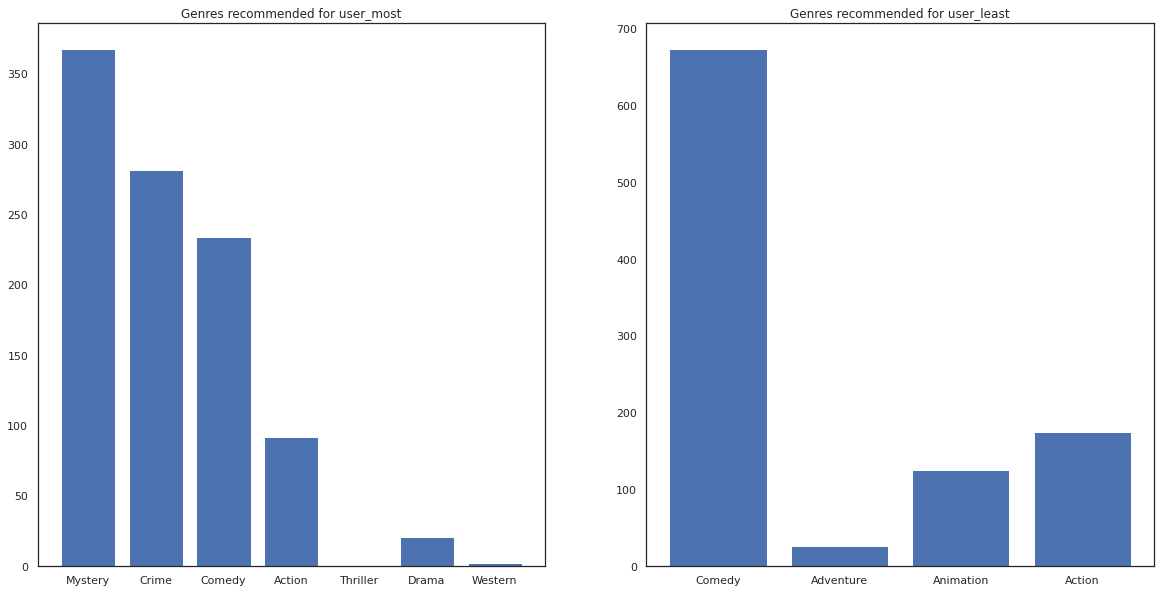
user_most ha visto un montón de películas y ha sido recomendado más géneros de nicho como el misterio y crimen mientras que user_least no ha visto muchas películas y se recomienda más películas de corriente, que la comedia y la acción de inclinación.
