 Ver en TensorFlow.org Ver en TensorFlow.org |  Ejecutar en Google Colab Ejecutar en Google Colab |  Ver en GitHub Ver en GitHub |  Descargar cuaderno Descargar cuaderno |  Ver modelo TF Hub Ver modelo TF Hub |
El cable de 19-texto giratorio módulo de TF-Hub incrustar ( https://tfhub.dev/tensorflow/cord-19/swivel-128d/3 ) fue construido para respaldar a los investigadores que analizan los lenguajes de texto naturales relacionados con COVID-19. Estas inclusiones fueron capacitados en los títulos, autores, resúmenes, textos del cuerpo, y los títulos de referencias de artículos en el conjunto de datos CORD-19 .
En este colab vamos a:
- Analizar palabras semánticamente similares en el espacio de inserción.
- Entrene un clasificador en el conjunto de datos de SciCite utilizando las incrustaciones de CORD-19
Configuración
import functools
import itertools
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import pandas as pd
import tensorflow as tf
import tensorflow_datasets as tfds
import tensorflow_hub as hub
from tqdm import trange
Analizar las incrustaciones
Comencemos analizando la incrustación calculando y trazando una matriz de correlación entre diferentes términos. Si la incrustación aprendió a capturar con éxito el significado de diferentes palabras, los vectores de incrustación de palabras semánticamente similares deberían estar muy juntos. Echemos un vistazo a algunos términos relacionados con COVID-19.
# Use the inner product between two embedding vectors as the similarity measure
def plot_correlation(labels, features):
corr = np.inner(features, features)
corr /= np.max(corr)
sns.heatmap(corr, xticklabels=labels, yticklabels=labels)
# Generate embeddings for some terms
queries = [
# Related viruses
'coronavirus', 'SARS', 'MERS',
# Regions
'Italy', 'Spain', 'Europe',
# Symptoms
'cough', 'fever', 'throat'
]
module = hub.load('https://tfhub.dev/tensorflow/cord-19/swivel-128d/3')
embeddings = module(queries)
plot_correlation(queries, embeddings)

Podemos ver que la incrustación capturó con éxito el significado de los diferentes términos. Cada palabra es similar a las otras palabras de su grupo (es decir, "coronavirus" tiene una alta correlación con "SARS" y "MERS"), mientras que son diferentes de los términos de otros grupos (es decir, la similitud entre "SARS" y "España" es cerca de 0).
Ahora veamos cómo podemos usar estas incrustaciones para resolver una tarea específica.
SciCite: Clasificación por intención de citas
Esta sección muestra cómo se puede utilizar la incrustación para tareas posteriores, como la clasificación de texto. Vamos a utilizar el conjunto de datos SciCite de TensorFlow conjuntos de datos a los intentos de citación classify de trabajos académicos. Dada una oración con una cita de un artículo académico, clasifique si la intención principal de la cita es como información de antecedentes, uso de métodos o comparación de resultados.
builder = tfds.builder(name='scicite')
builder.download_and_prepare()
train_data, validation_data, test_data = builder.as_dataset(
split=('train', 'validation', 'test'),
as_supervised=True)
Echemos un vistazo a algunos ejemplos etiquetados del conjunto de capacitación.
NUM_EXAMPLES = 10
TEXT_FEATURE_NAME = builder.info.supervised_keys[0]
LABEL_NAME = builder.info.supervised_keys[1]
def label2str(numeric_label):
m = builder.info.features[LABEL_NAME].names
return m[numeric_label]
data = next(iter(train_data.batch(NUM_EXAMPLES)))
pd.DataFrame({
TEXT_FEATURE_NAME: [ex.numpy().decode('utf8') for ex in data[0]],
LABEL_NAME: [label2str(x) for x in data[1]]
})
Entrenamiento de un clasificador de intenciones de citaton
Nos entrenaremos un clasificador en el conjunto de datos SciCite usando Keras. Construyamos un modelo que use las incrustaciones de CORD-19 con una capa de clasificación en la parte superior.
Hiperparámetros
EMBEDDING = 'https://tfhub.dev/tensorflow/cord-19/swivel-128d/3'
TRAINABLE_MODULE = False
hub_layer = hub.KerasLayer(EMBEDDING, input_shape=[],
dtype=tf.string, trainable=TRAINABLE_MODULE)
model = tf.keras.Sequential()
model.add(hub_layer)
model.add(tf.keras.layers.Dense(3))
model.summary()
model.compile(optimizer='adam',
loss=tf.keras.losses.SparseCategoricalCrossentropy(from_logits=True),
metrics=['accuracy'])
Model: "sequential"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
keras_layer (KerasLayer) (None, 128) 17301632
dense (Dense) (None, 3) 387
=================================================================
Total params: 17,302,019
Trainable params: 387
Non-trainable params: 17,301,632
_________________________________________________________________
Entrenar y evaluar el modelo
Entrenemos y evaluemos el modelo para ver el desempeño en la tarea SciCite
EPOCHS = 35
BATCH_SIZE = 32
history = model.fit(train_data.shuffle(10000).batch(BATCH_SIZE),
epochs=EPOCHS,
validation_data=validation_data.batch(BATCH_SIZE),
verbose=1)
Epoch 1/35 257/257 [==============================] - 3s 7ms/step - loss: 0.9244 - accuracy: 0.5924 - val_loss: 0.7915 - val_accuracy: 0.6627 Epoch 2/35 257/257 [==============================] - 2s 5ms/step - loss: 0.7097 - accuracy: 0.7152 - val_loss: 0.6799 - val_accuracy: 0.7358 Epoch 3/35 257/257 [==============================] - 2s 7ms/step - loss: 0.6317 - accuracy: 0.7551 - val_loss: 0.6285 - val_accuracy: 0.7544 Epoch 4/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5938 - accuracy: 0.7687 - val_loss: 0.6032 - val_accuracy: 0.7566 Epoch 5/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5724 - accuracy: 0.7750 - val_loss: 0.5871 - val_accuracy: 0.7653 Epoch 6/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5580 - accuracy: 0.7825 - val_loss: 0.5800 - val_accuracy: 0.7653 Epoch 7/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5484 - accuracy: 0.7870 - val_loss: 0.5711 - val_accuracy: 0.7718 Epoch 8/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5417 - accuracy: 0.7896 - val_loss: 0.5648 - val_accuracy: 0.7806 Epoch 9/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5356 - accuracy: 0.7902 - val_loss: 0.5628 - val_accuracy: 0.7740 Epoch 10/35 257/257 [==============================] - 2s 7ms/step - loss: 0.5313 - accuracy: 0.7903 - val_loss: 0.5581 - val_accuracy: 0.7849 Epoch 11/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5277 - accuracy: 0.7928 - val_loss: 0.5555 - val_accuracy: 0.7838 Epoch 12/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5242 - accuracy: 0.7940 - val_loss: 0.5528 - val_accuracy: 0.7849 Epoch 13/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5215 - accuracy: 0.7947 - val_loss: 0.5522 - val_accuracy: 0.7828 Epoch 14/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5190 - accuracy: 0.7961 - val_loss: 0.5527 - val_accuracy: 0.7751 Epoch 15/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5176 - accuracy: 0.7940 - val_loss: 0.5492 - val_accuracy: 0.7806 Epoch 16/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5154 - accuracy: 0.7978 - val_loss: 0.5500 - val_accuracy: 0.7817 Epoch 17/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5136 - accuracy: 0.7968 - val_loss: 0.5488 - val_accuracy: 0.7795 Epoch 18/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5127 - accuracy: 0.7967 - val_loss: 0.5504 - val_accuracy: 0.7838 Epoch 19/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5111 - accuracy: 0.7970 - val_loss: 0.5470 - val_accuracy: 0.7860 Epoch 20/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5101 - accuracy: 0.7972 - val_loss: 0.5471 - val_accuracy: 0.7871 Epoch 21/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5082 - accuracy: 0.7997 - val_loss: 0.5483 - val_accuracy: 0.7784 Epoch 22/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5077 - accuracy: 0.7995 - val_loss: 0.5471 - val_accuracy: 0.7860 Epoch 23/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5064 - accuracy: 0.8012 - val_loss: 0.5439 - val_accuracy: 0.7871 Epoch 24/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5057 - accuracy: 0.7990 - val_loss: 0.5476 - val_accuracy: 0.7882 Epoch 25/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5050 - accuracy: 0.7996 - val_loss: 0.5442 - val_accuracy: 0.7937 Epoch 26/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5045 - accuracy: 0.7999 - val_loss: 0.5455 - val_accuracy: 0.7860 Epoch 27/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5032 - accuracy: 0.7991 - val_loss: 0.5435 - val_accuracy: 0.7893 Epoch 28/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5034 - accuracy: 0.8022 - val_loss: 0.5431 - val_accuracy: 0.7882 Epoch 29/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5025 - accuracy: 0.8017 - val_loss: 0.5441 - val_accuracy: 0.7937 Epoch 30/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5017 - accuracy: 0.8013 - val_loss: 0.5463 - val_accuracy: 0.7838 Epoch 31/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5015 - accuracy: 0.8017 - val_loss: 0.5453 - val_accuracy: 0.7871 Epoch 32/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5011 - accuracy: 0.8014 - val_loss: 0.5448 - val_accuracy: 0.7915 Epoch 33/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5006 - accuracy: 0.8025 - val_loss: 0.5432 - val_accuracy: 0.7893 Epoch 34/35 257/257 [==============================] - 2s 5ms/step - loss: 0.5005 - accuracy: 0.8008 - val_loss: 0.5448 - val_accuracy: 0.7904 Epoch 35/35 257/257 [==============================] - 2s 5ms/step - loss: 0.4996 - accuracy: 0.8016 - val_loss: 0.5448 - val_accuracy: 0.7915
from matplotlib import pyplot as plt
def display_training_curves(training, validation, title, subplot):
if subplot%10==1: # set up the subplots on the first call
plt.subplots(figsize=(10,10), facecolor='#F0F0F0')
plt.tight_layout()
ax = plt.subplot(subplot)
ax.set_facecolor('#F8F8F8')
ax.plot(training)
ax.plot(validation)
ax.set_title('model '+ title)
ax.set_ylabel(title)
ax.set_xlabel('epoch')
ax.legend(['train', 'valid.'])
display_training_curves(history.history['accuracy'], history.history['val_accuracy'], 'accuracy', 211)
display_training_curves(history.history['loss'], history.history['val_loss'], 'loss', 212)
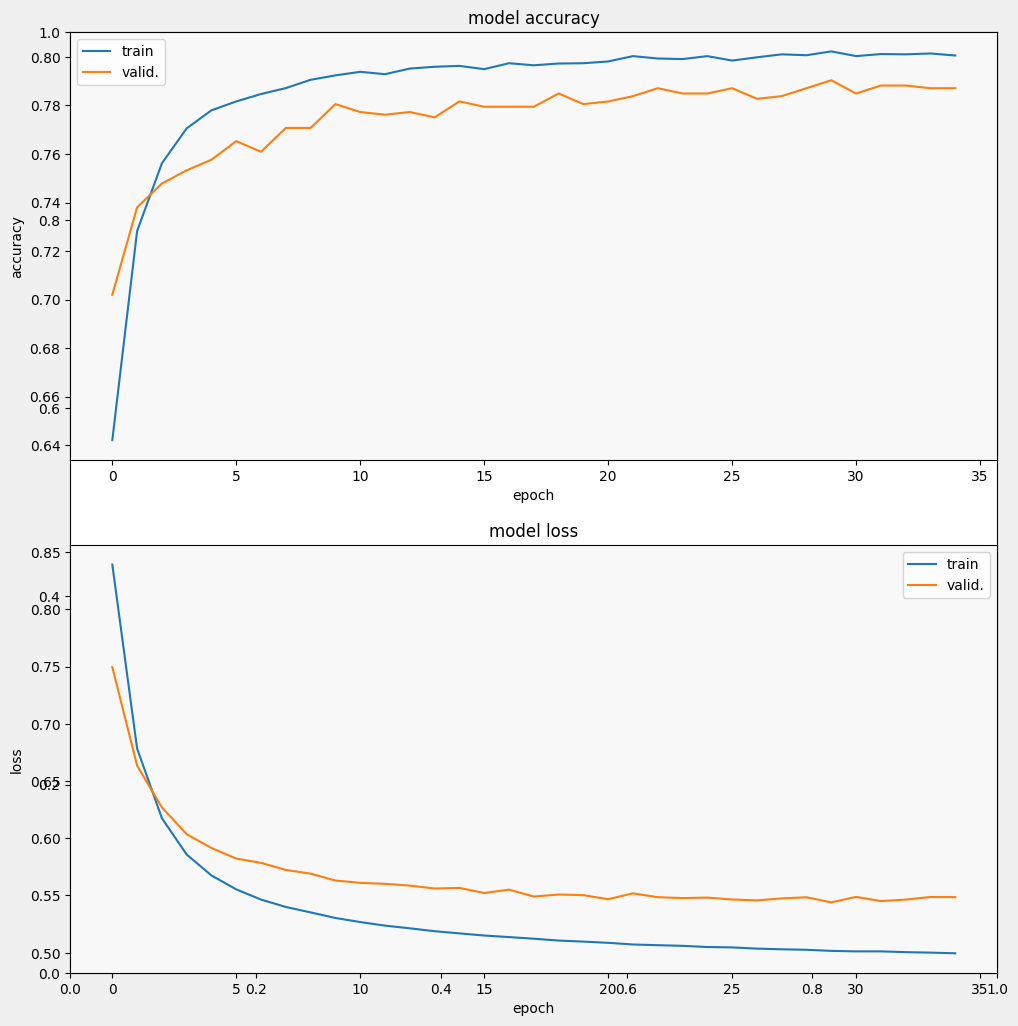
Evaluar el modelo
Y veamos cómo funciona el modelo. Se devolverán dos valores. Pérdida (un número que representa nuestro error, los valores más bajos son mejores) y precisión.
results = model.evaluate(test_data.batch(512), verbose=2)
for name, value in zip(model.metrics_names, results):
print('%s: %.3f' % (name, value))
4/4 - 0s - loss: 0.5357 - accuracy: 0.7891 - 441ms/epoch - 110ms/step loss: 0.536 accuracy: 0.789
Podemos ver que la pérdida disminuye rápidamente mientras que, especialmente, la precisión aumenta rápidamente. Tracemos algunos ejemplos para comprobar cómo se relaciona la predicción con las etiquetas verdaderas:
prediction_dataset = next(iter(test_data.batch(20)))
prediction_texts = [ex.numpy().decode('utf8') for ex in prediction_dataset[0]]
prediction_labels = [label2str(x) for x in prediction_dataset[1]]
predictions = [
label2str(x) for x in np.argmax(model.predict(prediction_texts), axis=-1)]
pd.DataFrame({
TEXT_FEATURE_NAME: prediction_texts,
LABEL_NAME: prediction_labels,
'prediction': predictions
})
Podemos ver que para esta muestra aleatoria, el modelo predice la etiqueta correcta la mayoría de las veces, lo que indica que puede incrustar oraciones científicas bastante bien.
¿Que sigue?
Ahora que ha aprendido un poco más sobre las incrustaciones CORD-19 Swivel de TF-Hub, lo alentamos a participar en la competencia CORD-19 Kaggle para contribuir a obtener conocimientos científicos de los textos académicos relacionados con COVID-19.
- Participar en la Kaggle Challenge CORD-19
- Más información sobre el COVID-19 Abierto de Investigación Conjunto de datos (CORD-19)
- Consulte la documentación y más acerca de las inclusiones TF-cubo en https://tfhub.dev/tensorflow/cord-19/swivel-128d/3
- Explora el espacio incrustación CORD-19 con la TensorFlow incrustación de proyector
