 Zobacz na TensorFlow.org Zobacz na TensorFlow.org |  Uruchom w Google Colab Uruchom w Google Colab |  Wyświetl źródło na GitHub Wyświetl źródło na GitHub |  Pobierz notatnik Pobierz notatnik |
Probabilistyczny Zasadnicza analiza komponentów (PCA) jest techniką redukcja wymiarów, które analizuje dane poprzez niższe wymiarowej przestrzeni utajonego ( Tipping i Bishop 1999 ). Jest często używany, gdy w danych brakuje wartości lub do skalowania wielowymiarowego.
Import
import functools
import warnings
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import tensorflow.compat.v2 as tf
import tensorflow_probability as tfp
from tensorflow_probability import bijectors as tfb
from tensorflow_probability import distributions as tfd
tf.enable_v2_behavior()
plt.style.use("ggplot")
warnings.filterwarnings('ignore')
Modelka
Rozważmy zbioru danych \(\mathbf{X} = \{\mathbf{x}_n\}\) z \(N\) punktów danych, przy czym każdy punkt pomiarowy \(D\)wymiarową, $ \ mathbf {x} _N \ in \ mathbb {R} ^ D\(. We aim to represent each \)\ mathbf {x} _N $ pod zmiennym utajony \(\mathbf{z}_n \in \mathbb{R}^K\) z dolnego wymiaru $ K <D\(. The set of principal axes \)\ mathbf {w} $ dotyczy zmienne ukryte w danych.
W szczególności zakładamy, że każda zmienna latentna ma rozkład normalny,
\[ \begin{equation*} \mathbf{z}_n \sim N(\mathbf{0}, \mathbf{I}). \end{equation*} \]
Odpowiedni punkt danych jest generowany przez projekcję,
\[ \begin{equation*} \mathbf{x}_n \mid \mathbf{z}_n \sim N(\mathbf{W}\mathbf{z}_n, \sigma^2\mathbf{I}), \end{equation*} \]
gdzie macierz \(\mathbf{W}\in\mathbb{R}^{D\times K}\) zwanych głównymi osiami. W probabilistyczny PCA, jesteśmy zazwyczaj zainteresowany szacowania osi głównych \(\mathbf{W}\) a termin hałas\(\sigma^2\).
Probabilistyczny PCA uogólnia klasyczny PCA. Marginalizując zmienną ukrytą, rozkład każdego punktu danych wynosi
\[ \begin{equation*} \mathbf{x}_n \sim N(\mathbf{0}, \mathbf{W}\mathbf{W}^\top + \sigma^2\mathbf{I}). \end{equation*} \]
PCA jest klasyczny przypadek specyficzny probabilistycznego PCA gdy kowariancja szumu staje się nieskończenie mały, \(\sigma^2 \to 0\).
Poniżej przedstawiamy nasz model. W naszej analizie zakładamy \(\sigma\) jest znana, a zamiast punktu szacowania \(\mathbf{W}\) jako parametr modelu kładziemy uprzednie nad nim, aby wyprowadzić rozkład na głównych osiach. Będziemy wyrazić model jako TFP JointDistribution konkretnie użyjemy JointDistributionCoroutineAutoBatched .
def probabilistic_pca(data_dim, latent_dim, num_datapoints, stddv_datapoints):
w = yield tfd.Normal(loc=tf.zeros([data_dim, latent_dim]),
scale=2.0 * tf.ones([data_dim, latent_dim]),
name="w")
z = yield tfd.Normal(loc=tf.zeros([latent_dim, num_datapoints]),
scale=tf.ones([latent_dim, num_datapoints]),
name="z")
x = yield tfd.Normal(loc=tf.matmul(w, z),
scale=stddv_datapoints,
name="x")
num_datapoints = 5000
data_dim = 2
latent_dim = 1
stddv_datapoints = 0.5
concrete_ppca_model = functools.partial(probabilistic_pca,
data_dim=data_dim,
latent_dim=latent_dim,
num_datapoints=num_datapoints,
stddv_datapoints=stddv_datapoints)
model = tfd.JointDistributionCoroutineAutoBatched(concrete_ppca_model)
Dane
Możemy użyć modelu do wygenerowania danych przez próbkowanie ze wspólnego wcześniejszego rozkładu.
actual_w, actual_z, x_train = model.sample()
print("Principal axes:")
print(actual_w)
Principal axes: tf.Tensor( [[ 2.2801023] [-1.1619819]], shape=(2, 1), dtype=float32)
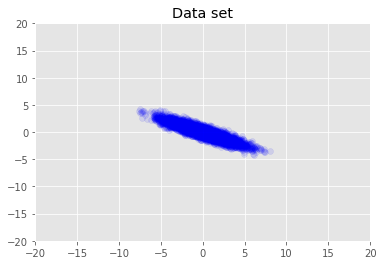
Wizualizujemy zbiór danych.
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1)
plt.axis([-20, 20, -20, 20])
plt.title("Data set")
plt.show()
Maksimum a posteriori wnioskowanie
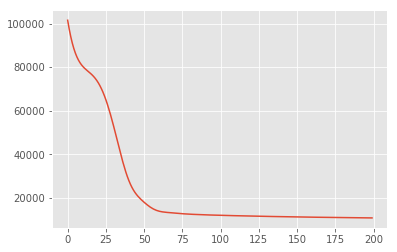
Najpierw szukamy oszacowania punktowego zmiennych latentnych, które maksymalizuje gęstość prawdopodobieństwa a posteriori. Jest to znane jako maksimum a posteriori (MAP) wnioskowanie, i odbywa się przez obliczenie wartości \(\mathbf{W}\) i \(\mathbf{Z}\) które maksymalizują tylną gęstości \(p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X}) \propto p(\mathbf{W}, \mathbf{Z}, \mathbf{X})\).
w = tf.Variable(tf.random.normal([data_dim, latent_dim]))
z = tf.Variable(tf.random.normal([latent_dim, num_datapoints]))
target_log_prob_fn = lambda w, z: model.log_prob((w, z, x_train))
losses = tfp.math.minimize(
lambda: -target_log_prob_fn(w, z),
optimizer=tf.optimizers.Adam(learning_rate=0.05),
num_steps=200)
plt.plot(losses)
[<matplotlib.lines.Line2D at 0x7f19897a42e8>]
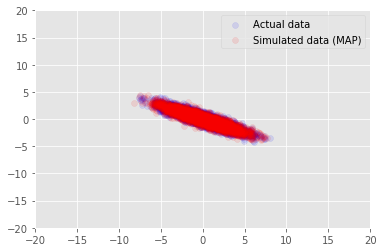
Możemy wykorzystać model do przykładowych danych dla wnioskować wartościami \(\mathbf{W}\) i \(\mathbf{Z}\)i porównać z rzeczywistym zbiorze mamy uzależnionych.
print("MAP-estimated axes:")
print(w)
_, _, x_generated = model.sample(value=(w, z, None))
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1, label='Actual data')
plt.scatter(x_generated[0, :], x_generated[1, :], color='red', alpha=0.1, label='Simulated data (MAP)')
plt.legend()
plt.axis([-20, 20, -20, 20])
plt.show()
MAP-estimated axes:
<tf.Variable 'Variable:0' shape=(2, 1) dtype=float32, numpy=
array([[ 2.9135954],
[-1.4826864]], dtype=float32)>
Wnioskowanie wariacyjne
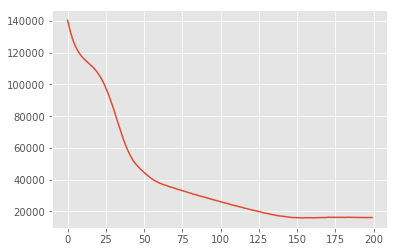
MAP może być użyty do znalezienia modu (lub jednego z modów) rozkładu a posteriori, ale nie dostarcza żadnych innych informacji na ten temat. Mamy kolejny użyć wariacyjne wnioskowanie, w którym tylna distribtion \(p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X})\) jest przybliżeniem za pomocą dystrybucji wariacyjna \(q(\mathbf{W}, \mathbf{Z})\) sparametryzowane przez \(\boldsymbol{\lambda}\). Celem jest znalezienie parametrów wariacyjnych \(\boldsymbol{\lambda}\) że zminimalizowania rozbieżności między q KL i tylnej, \(\mathrm{KL}(q(\mathbf{W}, \mathbf{Z}) \mid\mid p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X}))\)lub równoważnie, które maksymalizują dowodów dolna granica, \(\mathbb{E}_{q(\mathbf{W},\mathbf{Z};\boldsymbol{\lambda})}\left[ \log p(\mathbf{W},\mathbf{Z},\mathbf{X}) - \log q(\mathbf{W},\mathbf{Z}; \boldsymbol{\lambda}) \right]\).
qw_mean = tf.Variable(tf.random.normal([data_dim, latent_dim]))
qz_mean = tf.Variable(tf.random.normal([latent_dim, num_datapoints]))
qw_stddv = tfp.util.TransformedVariable(1e-4 * tf.ones([data_dim, latent_dim]),
bijector=tfb.Softplus())
qz_stddv = tfp.util.TransformedVariable(
1e-4 * tf.ones([latent_dim, num_datapoints]),
bijector=tfb.Softplus())
def factored_normal_variational_model():
qw = yield tfd.Normal(loc=qw_mean, scale=qw_stddv, name="qw")
qz = yield tfd.Normal(loc=qz_mean, scale=qz_stddv, name="qz")
surrogate_posterior = tfd.JointDistributionCoroutineAutoBatched(
factored_normal_variational_model)
losses = tfp.vi.fit_surrogate_posterior(
target_log_prob_fn,
surrogate_posterior=surrogate_posterior,
optimizer=tf.optimizers.Adam(learning_rate=0.05),
num_steps=200)
print("Inferred axes:")
print(qw_mean)
print("Standard Deviation:")
print(qw_stddv)
plt.plot(losses)
plt.show()
Inferred axes:
<tf.Variable 'Variable:0' shape=(2, 1) dtype=float32, numpy=
array([[ 2.4168603],
[-1.2236133]], dtype=float32)>
Standard Deviation:
<TransformedVariable: dtype=float32, shape=[2, 1], fn="softplus", numpy=
array([[0.0042499 ],
[0.00598824]], dtype=float32)>
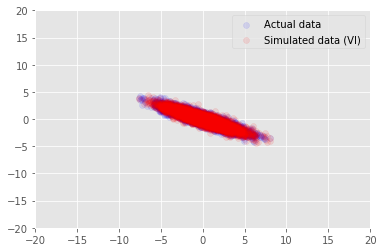
posterior_samples = surrogate_posterior.sample(50)
_, _, x_generated = model.sample(value=(posterior_samples))
# It's a pain to plot all 5000 points for each of our 50 posterior samples, so
# let's subsample to get the gist of the distribution.
x_generated = tf.reshape(tf.transpose(x_generated, [1, 0, 2]), (2, -1))[:, ::47]
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1, label='Actual data')
plt.scatter(x_generated[0, :], x_generated[1, :], color='red', alpha=0.1, label='Simulated data (VI)')
plt.legend()
plt.axis([-20, 20, -20, 20])
plt.show()
Podziękowanie
W Edward 1.0 (Ten poradnik został napisany źródłowego ). Dziękujemy wszystkim współtwórcom za napisanie i poprawienie tej wersji.
Bibliografia
[1]: Michael E. Napiwki i Christopher M. Bishop. Analiza probabilistyczna głównych składowych. Journal of Royal Society Statystycznego: Seria B (metoda statystyczna), 61 (3): 611-622, 1999.
