 عرض على TensorFlow.org عرض على TensorFlow.org |  تشغيل في Google Colab تشغيل في Google Colab |  عرض المصدر على جيثب عرض المصدر على جيثب |  تحميل دفتر تحميل دفتر |
في هذا colab ، سنستكشف أخذ العينات من الجزء الخلفي من نموذج خليط غاوسي بايزي (BGMM) باستخدام العناصر الأولية الاحتمالية لـ TensorFlow فقط.
نموذج
ل \(k\in\{1,\ldots, K\}\) مكونات الخليط كل البعد \(D\)، نود أن نموذج \(i\in\{1,\ldots,N\}\) IID العينات باستخدام ما يلي النظرية الافتراضية التمويه خليط نموذج:
\[\begin{align*} \theta &\sim \text{Dirichlet}(\text{concentration}=\alpha_0)\\ \mu_k &\sim \text{Normal}(\text{loc}=\mu_{0k}, \text{scale}=I_D)\\ T_k &\sim \text{Wishart}(\text{df}=5, \text{scale}=I_D)\\ Z_i &\sim \text{Categorical}(\text{probs}=\theta)\\ Y_i &\sim \text{Normal}(\text{loc}=\mu_{z_i}, \text{scale}=T_{z_i}^{-1/2})\\ \end{align*}\]
ملاحظة، و scale الحجج يكون كل cholesky دلالات. نحن نستخدم هذه الاتفاقية لأنها خاصة بتوزيعات TF (التي تستخدم هذه الاتفاقية في حد ذاتها لأنها مفيدة من الناحية الحسابية).
هدفنا هو توليد عينات من الخلف:
\[p\left(\theta, \{\mu_k, T_k\}_{k=1}^K \Big| \{y_i\}_{i=1}^N, \alpha_0, \{\mu_{ok}\}_{k=1}^K\right)\]
لاحظ أن \(\{Z_i\}_{i=1}^N\) غير موجود - we're المهتمة في فقط تلك المتغيرات العشوائية التي لا النطاق مع \(N\). (ولحسن الحظ هناك توزيع TF الذي يعالج تهميش من \(Z_i\)).
ليس من الممكن أخذ عينة مباشرة من هذا التوزيع بسبب مصطلح تطبيع حسابي صعب الحل.
خوارزميات المدينة، هاستينغز هي تقنية للأخذ عينات من توزيعات-المستعصية إلى تطبيع.
تقدم TensorFlow Probability عددًا من خيارات MCMC ، بما في ذلك العديد منها استنادًا إلى Metropolis-Hastings. في هذه المفكرة، سنستخدم هاملتون مونت كارلو ( tfp.mcmc.HamiltonianMonteCarlo ). غالبًا ما تكون HMC خيارًا جيدًا لأنها يمكن أن تتقارب بسرعة ، وتختبر مساحة الدولة بشكل مشترك (على عكس التنسيق) ، وتعزز إحدى فضائل TF: التمايز التلقائي. وقال، وأخذ العينات من الخلفي BGMM قد فعلا القيام به بشكل أفضل من خلال المناهج الأخرى، على سبيل المثال، أخذ العينات جيب ل .
%matplotlib inline
import functools
import matplotlib.pyplot as plt; plt.style.use('ggplot')
import numpy as np
import seaborn as sns; sns.set_context('notebook')
import tensorflow.compat.v2 as tf
tf.enable_v2_behavior()
import tensorflow_probability as tfp
tfd = tfp.distributions
tfb = tfp.bijectors
physical_devices = tf.config.experimental.list_physical_devices('GPU')
if len(physical_devices) > 0:
tf.config.experimental.set_memory_growth(physical_devices[0], True)
قبل إنشاء النموذج فعليًا ، سنحتاج إلى تحديد نوع جديد من التوزيع. من مواصفات النموذج أعلاه ، من الواضح أننا نضع معلمات لـ MVN بمصفوفة تغاير معكوسة ، على سبيل المثال ، [مصفوفة الدقة] (https://en.wikipedia.org/wiki/Precision_ (الإحصائيات٪ 29). لتحقيق ذلك في TF، سوف نحتاج إلى طرح دينا Bijector هذا. Bijector سوف تستخدم التحول إلى الأمام:
-
Y = tf.linalg.triangular_solve((tf.linalg.matrix_transpose(chol_precision_tril), X, adjoint=True) + loc.
و log_prob الحساب هو فقط معكوس، أي:
-
X = tf.linalg.matmul(chol_precision_tril, X - loc, adjoint_a=True).
وبما أن كل حاجة لدينا لمؤسسة حمد الطبية هي log_prob ، يعني ذلك أن نتجنب أي وقت مضى داعيا tf.linalg.triangular_solve (كما هو الحال ل tfd.MultivariateNormalTriL ). هذا هو مفيد منذ tf.linalg.matmul عادة أسرع المستحقة لأفضل موقع ذاكرة التخزين المؤقت.
class MVNCholPrecisionTriL(tfd.TransformedDistribution):
"""MVN from loc and (Cholesky) precision matrix."""
def __init__(self, loc, chol_precision_tril, name=None):
super(MVNCholPrecisionTriL, self).__init__(
distribution=tfd.Independent(tfd.Normal(tf.zeros_like(loc),
scale=tf.ones_like(loc)),
reinterpreted_batch_ndims=1),
bijector=tfb.Chain([
tfb.Shift(shift=loc),
tfb.Invert(tfb.ScaleMatvecTriL(scale_tril=chol_precision_tril,
adjoint=True)),
]),
name=name)
و tfd.Independent توجه مستقلة يتحول توزيع توزيع واحدة، في توزيع متعدد المتغيرات مع الإحداثيات مستقلة إحصائيا. من حيث الحوسبة log_prob ، وهذا "الفوقية توزيع" يتظاهر في شكل مبلغ بسيط على البعد الحدث (ق).
ولاحظ أيضا أن اتخذنا adjoint ( "تبديل") من مصفوفة الحجم. هذا هو لأنه إذا الدقة هو التغاير معكوس، أي \(P=C^{-1}\) وإذا \(C=AA^\top\)، ثم \(P=BB^{\top}\) حيث \(B=A^{-\top}\).
وبما أن هذا التوزيع هو نوع من صعبة، دعونا تحقق بسرعة أن لدينا MVNCholPrecisionTriL يعمل كما نعتقد أنه ينبغي.
def compute_sample_stats(d, seed=42, n=int(1e6)):
x = d.sample(n, seed=seed)
sample_mean = tf.reduce_mean(x, axis=0, keepdims=True)
s = x - sample_mean
sample_cov = tf.linalg.matmul(s, s, adjoint_a=True) / tf.cast(n, s.dtype)
sample_scale = tf.linalg.cholesky(sample_cov)
sample_mean = sample_mean[0]
return [
sample_mean,
sample_cov,
sample_scale,
]
dtype = np.float32
true_loc = np.array([1., -1.], dtype=dtype)
true_chol_precision = np.array([[1., 0.],
[2., 8.]],
dtype=dtype)
true_precision = np.matmul(true_chol_precision, true_chol_precision.T)
true_cov = np.linalg.inv(true_precision)
d = MVNCholPrecisionTriL(
loc=true_loc,
chol_precision_tril=true_chol_precision)
[sample_mean, sample_cov, sample_scale] = [
t.numpy() for t in compute_sample_stats(d)]
print('true mean:', true_loc)
print('sample mean:', sample_mean)
print('true cov:\n', true_cov)
print('sample cov:\n', sample_cov)
true mean: [ 1. -1.] sample mean: [ 1.0002806 -1.000105 ] true cov: [[ 1.0625 -0.03125 ] [-0.03125 0.015625]] sample cov: [[ 1.0641273 -0.03126175] [-0.03126175 0.01559312]]
نظرًا لأن متوسط العينة والتغاير قريبان من المتوسط الحقيقي والتغاير ، يبدو أن التوزيع يتم تنفيذه بشكل صحيح. الآن، سنستخدم MVNCholPrecisionTriL tfp.distributions.JointDistributionNamed لتحديد نموذج BGMM. لنموذج الرصد، سنستخدم tfd.MixtureSameFamily لدمج تلقائيا خارج \(\{Z_i\}_{i=1}^N\) تعادلات.
dtype = np.float64
dims = 2
components = 3
num_samples = 1000
bgmm = tfd.JointDistributionNamed(dict(
mix_probs=tfd.Dirichlet(
concentration=np.ones(components, dtype) / 10.),
loc=tfd.Independent(
tfd.Normal(
loc=np.stack([
-np.ones(dims, dtype),
np.zeros(dims, dtype),
np.ones(dims, dtype),
]),
scale=tf.ones([components, dims], dtype)),
reinterpreted_batch_ndims=2),
precision=tfd.Independent(
tfd.WishartTriL(
df=5,
scale_tril=np.stack([np.eye(dims, dtype=dtype)]*components),
input_output_cholesky=True),
reinterpreted_batch_ndims=1),
s=lambda mix_probs, loc, precision: tfd.Sample(tfd.MixtureSameFamily(
mixture_distribution=tfd.Categorical(probs=mix_probs),
components_distribution=MVNCholPrecisionTriL(
loc=loc,
chol_precision_tril=precision)),
sample_shape=num_samples)
))
def joint_log_prob(observations, mix_probs, loc, chol_precision):
"""BGMM with priors: loc=Normal, precision=Inverse-Wishart, mix=Dirichlet.
Args:
observations: `[n, d]`-shaped `Tensor` representing Bayesian Gaussian
Mixture model draws. Each sample is a length-`d` vector.
mix_probs: `[K]`-shaped `Tensor` representing random draw from
`Dirichlet` prior.
loc: `[K, d]`-shaped `Tensor` representing the location parameter of the
`K` components.
chol_precision: `[K, d, d]`-shaped `Tensor` representing `K` lower
triangular `cholesky(Precision)` matrices, each being sampled from
a Wishart distribution.
Returns:
log_prob: `Tensor` representing joint log-density over all inputs.
"""
return bgmm.log_prob(
mix_probs=mix_probs, loc=loc, precision=chol_precision, s=observations)
توليد بيانات "تدريب"
في هذا العرض التوضيحي ، سنقوم بأخذ عينات من بعض البيانات العشوائية.
true_loc = np.array([[-2., -2],
[0, 0],
[2, 2]], dtype)
random = np.random.RandomState(seed=43)
true_hidden_component = random.randint(0, components, num_samples)
observations = (true_loc[true_hidden_component] +
random.randn(num_samples, dims).astype(dtype))
الاستدلال بايزي باستخدام HMC
الآن بعد أن استخدمنا TFD لتحديد نموذجنا وحصلنا على بعض البيانات المرصودة ، لدينا جميع القطع اللازمة لتشغيل HMC.
للقيام بذلك، ونحن سوف تستخدم التطبيق الجزئي ل"دبوس أسفل" الأشياء التي لا تريد أن العينة. في هذه الحالة هذا يعني أننا بحاجة دبوس فقط بانخفاض observations . (والمخبوزات والمعلمات فرط بالفعل إلى التوزيعات السابقة وليس جزءا من joint_log_prob ظيفة التوقيع).
unnormalized_posterior_log_prob = functools.partial(joint_log_prob, observations)
initial_state = [
tf.fill([components],
value=np.array(1. / components, dtype),
name='mix_probs'),
tf.constant(np.array([[-2., -2],
[0, 0],
[2, 2]], dtype),
name='loc'),
tf.linalg.eye(dims, batch_shape=[components], dtype=dtype, name='chol_precision'),
]
التمثيل غير المقيد
يتطلب هاميلتونيان مونت كارلو (HMC) أن تكون دالة احتمالية السجل المستهدفة قابلة للتفاضل فيما يتعلق بالحجج الخاصة بها. علاوة على ذلك ، يمكن لمؤسسة HMC إظهار كفاءة إحصائية أعلى بشكل كبير إذا كانت مساحة الدولة غير مقيدة.
هذا يعني أنه سيتعين علينا حل مشكلتين رئيسيتين عند أخذ العينات من BGMM اللاحق:
- \(\theta\) يمثل ناقلات احتمال منفصلة، أي يجب أن يكون مثل هذا \(\sum_{k=1}^K \theta_k = 1\) و \(\theta_k>0\).
- \(T_k\) يمثل مصفوفة، أي يجب أن يكون مثل أن معكوس التغاير \(T_k \succ 0\)، أي هو إيجابي واضح .
لتلبية هذا المطلب ، سنحتاج إلى:
- تحويل المتغيرات المقيدة إلى مساحة غير مقيدة
- قم بتشغيل MCMC في مساحة غير مقيدة
- تحويل المتغيرات غير المقيدة إلى المساحة المقيدة.
كما هو الحال مع MVNCholPrecisionTriL ، سنستخدم Bijector الصورة لتحويل المتغيرات العشوائية إلى الفضاء غير المقيد.
و
Dirichletتتحول إلى الفضاء غير المقيد عن طريق وظيفة softmax .المتغير العشوائي الدقيق الخاص بنا هو توزيع على المصفوفات شبه المحددة. لإلغاء تقييد هذه سنستخدم
FillTriangularوTransformDiagonalbijectors. تقوم هذه المتجهات بتحويل المصفوفات المثلثية السفلية وتضمن أن يكون القطر موجبًا. الأول هو مفيد لأنه تمكن أخذ العينات فقط \(d(d+1)/2\) يطفو بدلا من \(d^2\).
unconstraining_bijectors = [
tfb.SoftmaxCentered(),
tfb.Identity(),
tfb.Chain([
tfb.TransformDiagonal(tfb.Softplus()),
tfb.FillTriangular(),
])]
@tf.function(autograph=False)
def sample():
return tfp.mcmc.sample_chain(
num_results=2000,
num_burnin_steps=500,
current_state=initial_state,
kernel=tfp.mcmc.SimpleStepSizeAdaptation(
tfp.mcmc.TransformedTransitionKernel(
inner_kernel=tfp.mcmc.HamiltonianMonteCarlo(
target_log_prob_fn=unnormalized_posterior_log_prob,
step_size=0.065,
num_leapfrog_steps=5),
bijector=unconstraining_bijectors),
num_adaptation_steps=400),
trace_fn=lambda _, pkr: pkr.inner_results.inner_results.is_accepted)
[mix_probs, loc, chol_precision], is_accepted = sample()
سنقوم الآن بتنفيذ السلسلة وطباعة الوسيلة اللاحقة.
acceptance_rate = tf.reduce_mean(tf.cast(is_accepted, dtype=tf.float32)).numpy()
mean_mix_probs = tf.reduce_mean(mix_probs, axis=0).numpy()
mean_loc = tf.reduce_mean(loc, axis=0).numpy()
mean_chol_precision = tf.reduce_mean(chol_precision, axis=0).numpy()
precision = tf.linalg.matmul(chol_precision, chol_precision, transpose_b=True)
print('acceptance_rate:', acceptance_rate)
print('avg mix probs:', mean_mix_probs)
print('avg loc:\n', mean_loc)
print('avg chol(precision):\n', mean_chol_precision)
acceptance_rate: 0.5305 avg mix probs: [0.25248723 0.60729516 0.1402176 ] avg loc: [[-1.96466753 -2.12047249] [ 0.27628865 0.22944732] [ 2.06461244 2.54216122]] avg chol(precision): [[[ 1.05105032 0. ] [ 0.12699955 1.06553113]] [[ 0.76058015 0. ] [-0.50332767 0.77947431]] [[ 1.22770457 0. ] [ 0.70670027 1.50914164]]]
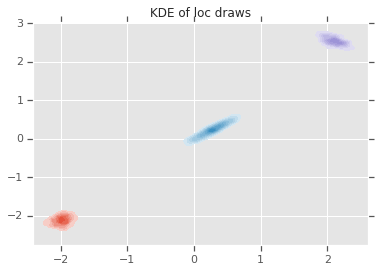
loc_ = loc.numpy()
ax = sns.kdeplot(loc_[:,0,0], loc_[:,0,1], shade=True, shade_lowest=False)
ax = sns.kdeplot(loc_[:,1,0], loc_[:,1,1], shade=True, shade_lowest=False)
ax = sns.kdeplot(loc_[:,2,0], loc_[:,2,1], shade=True, shade_lowest=False)
plt.title('KDE of loc draws');
استنتاج
أوضح هذا colab البسيط كيف يمكن استخدام بدائل TensorFlow الاحتمالية لبناء نماذج خليط بايزي الهرمية.
