 TensorFlow.org'da görüntüleyin TensorFlow.org'da görüntüleyin |  Google Colab'da çalıştırın Google Colab'da çalıştırın |  Kaynağı GitHub'da görüntüleyin Kaynağı GitHub'da görüntüleyin |  Not defterini indir Not defterini indir |
Sekiz okullar sorunu ( 1981 Rubin ) sekiz okulda paralel olarak yürütülen SAT koçluk programlarının etkinliğini değerlendirmektedir. Bu klasik bir sorunu (haline gelmiştir Bayes Veri Analizi , Stan değiştirilebilir gruplar arasında bilgi paylaşımı için hiyerarşik modelleme yararlılığını göstermektedir).
Aşağıdaki Gerçekleştiren Bir Edward 1.0 bir uyarlamasıdır öğretici .
ithalat
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import tensorflow.compat.v2 as tf
import tensorflow_probability as tfp
from tensorflow_probability import distributions as tfd
import warnings
tf.enable_v2_behavior()
plt.style.use("ggplot")
warnings.filterwarnings('ignore')
Veri
Bayesian Veri Analizi, bölüm 5.5'ten (Gelman ve diğerleri. 2013):
Sekiz lisenin her birinde SAT-V (Scholastic Aptitude Test-Verbal) için özel koçluk programlarının etkilerini analiz etmek için Educational Testing Service için bir çalışma yapıldı. Her çalışmadaki sonuç değişkeni, Educational Testing Service tarafından yönetilen ve kolejlerin kabul kararlarını vermelerine yardımcı olmak için kullanılan standart bir çoktan seçmeli test olan SAT-V'nin özel bir idaresindeki puandı; puanlar ortalama 500 ve standart sapma 100 civarında olmak üzere 200 ile 800 arasında değişebilir. SAT sınavları, özellikle testteki performansı iyileştirmeye yönelik kısa vadeli çabalara dirençli olacak şekilde tasarlanmıştır; bunun yerine, uzun yıllar eğitimle kazanılan bilgileri ve geliştirilen yetenekleri yansıtacak şekilde tasarlanmıştır. Bununla birlikte, bu çalışmadaki sekiz okulun her biri, kısa vadeli koçluk programının SAT puanlarını artırmada çok başarılı olduğunu düşündü. Ayrıca, sekiz programdan herhangi birinin diğerinden daha etkili olduğuna veya bazılarının etki bakımından diğerlerinden daha benzer olduğuna inanmak için önceden hiçbir neden yoktu.
Sekiz okulların (her biri için\(J = 8\)), biz tahmini tedavi etkisi var \(y_j\) ve efekt tahmini standart hatası \(\sigma_j\). Çalışmadaki tedavi etkileri, kontrol değişkenleri olarak PSAT-M ve PSAT-V puanları kullanılarak tedavi grubu üzerinde doğrusal bir regresyon ile elde edilmiştir. Okulların daha az veya benzer veya koçluk programlarından birini daha etkili olacağını olduklarını önceden hiçbir inanç yoktu, biz olarak tedavi etkilerini düşünebiliriz değiştirilebilir .
num_schools = 8 # number of schools
treatment_effects = np.array(
[28, 8, -3, 7, -1, 1, 18, 12], dtype=np.float32) # treatment effects
treatment_stddevs = np.array(
[15, 10, 16, 11, 9, 11, 10, 18], dtype=np.float32) # treatment SE
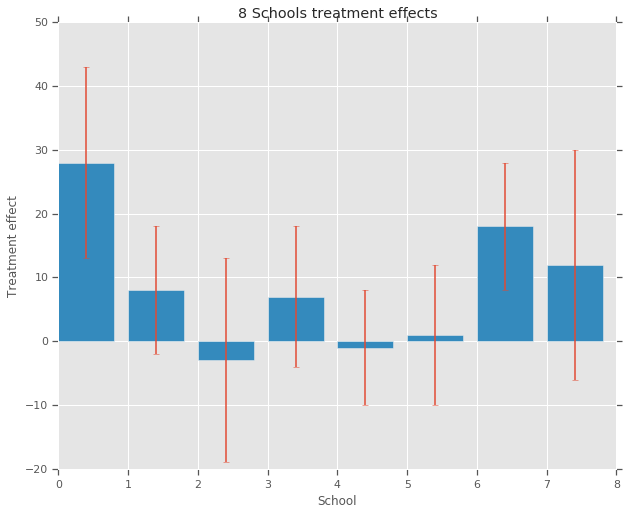
fig, ax = plt.subplots()
plt.bar(range(num_schools), treatment_effects, yerr=treatment_stddevs)
plt.title("8 Schools treatment effects")
plt.xlabel("School")
plt.ylabel("Treatment effect")
fig.set_size_inches(10, 8)
plt.show()
modeli
Verileri yakalamak için hiyerarşik bir normal model kullanıyoruz. Üretken süreci takip eder,
\[ \begin{align*} \mu &\sim \text{Normal}(\text{loc}{=}0,\ \text{scale}{=}10) \\ \log\tau &\sim \text{Normal}(\text{loc}{=}5,\ \text{scale}{=}1) \\ \text{for } & i=1\ldots 8:\\ & \theta_i \sim \text{Normal}\left(\text{loc}{=}\mu,\ \text{scale}{=}\tau \right) \\ & y_i \sim \text{Normal}\left(\text{loc}{=}\theta_i,\ \text{scale}{=}\sigma_i \right) \end{align*} \]
nerede \(\mu\) öncesinde ortalama tedavi etkisi ve temsil \(\tau\) kontrollerini ne kadar varyans okullar arasında yoktur. \(y_i\) ve \(\sigma_i\) gözlenir. As \(\tau \rightarrow \infty\), model okul tedavi etkisi tahminleri birbirinden bağımsız olmalarına izin verilen, yani no-havuzlama modelini yaklaşır. As \(\tau \rightarrow 0\), model okul tedavi etkilerinin tüm yakın grup ortalama üzeresiniz, yani tam-havuzlama modeli yaklaşımlar \(\mu\). Pozitif olduğu standart sapma sınırlamak için, çekme \(\tau\) (çizim eşdeğer bir lognormal dağılım gelen \(log(\tau)\) normal dağılımdan).
Aşağıdaki sapma ile Teşhis Sapmalı Çıkarım biz eşdeğer olmayan merkezli modele yukardaki modeli dönüşümü:
\[ \begin{align*} \mu &\sim \text{Normal}(\text{loc}{=}0,\ \text{scale}{=}10) \\ \log\tau &\sim \text{Normal}(\text{loc}{=}5,\ \text{scale}{=}1) \\ \text{for } & i=1\ldots 8:\\ & \theta_i' \sim \text{Normal}\left(\text{loc}{=}0,\ \text{scale}{=}1 \right) \\ & \theta_i = \mu + \tau \theta_i' \\ & y_i \sim \text{Normal}\left(\text{loc}{=}\theta_i,\ \text{scale}{=}\sigma_i \right) \end{align*} \]
Biz bu modeli reify JointDistributionSequential örneği:
model = tfd.JointDistributionSequential([
tfd.Normal(loc=0., scale=10., name="avg_effect"), # `mu` above
tfd.Normal(loc=5., scale=1., name="avg_stddev"), # `log(tau)` above
tfd.Independent(tfd.Normal(loc=tf.zeros(num_schools),
scale=tf.ones(num_schools),
name="school_effects_standard"), # `theta_prime`
reinterpreted_batch_ndims=1),
lambda school_effects_standard, avg_stddev, avg_effect: (
tfd.Independent(tfd.Normal(loc=(avg_effect[..., tf.newaxis] +
tf.exp(avg_stddev[..., tf.newaxis]) *
school_effects_standard), # `theta` above
scale=treatment_stddevs),
name="treatment_effects", # `y` above
reinterpreted_batch_ndims=1))
])
def target_log_prob_fn(avg_effect, avg_stddev, school_effects_standard):
"""Unnormalized target density as a function of states."""
return model.log_prob((
avg_effect, avg_stddev, school_effects_standard, treatment_effects))
Bayes Çıkarımı
Verilen veriler, modelin parametreleri üzerindeki sonsal dağılımı hesaplamak için Hamilton Monte Carlo (HMC) uyguluyoruz.
num_results = 5000
num_burnin_steps = 3000
# Improve performance by tracing the sampler using `tf.function`
# and compiling it using XLA.
@tf.function(autograph=False, jit_compile=True)
def do_sampling():
return tfp.mcmc.sample_chain(
num_results=num_results,
num_burnin_steps=num_burnin_steps,
current_state=[
tf.zeros([], name='init_avg_effect'),
tf.zeros([], name='init_avg_stddev'),
tf.ones([num_schools], name='init_school_effects_standard'),
],
kernel=tfp.mcmc.HamiltonianMonteCarlo(
target_log_prob_fn=target_log_prob_fn,
step_size=0.4,
num_leapfrog_steps=3))
states, kernel_results = do_sampling()
avg_effect, avg_stddev, school_effects_standard = states
school_effects_samples = (
avg_effect[:, np.newaxis] +
np.exp(avg_stddev)[:, np.newaxis] * school_effects_standard)
num_accepted = np.sum(kernel_results.is_accepted)
print('Acceptance rate: {}'.format(num_accepted / num_results))
Acceptance rate: 0.5974
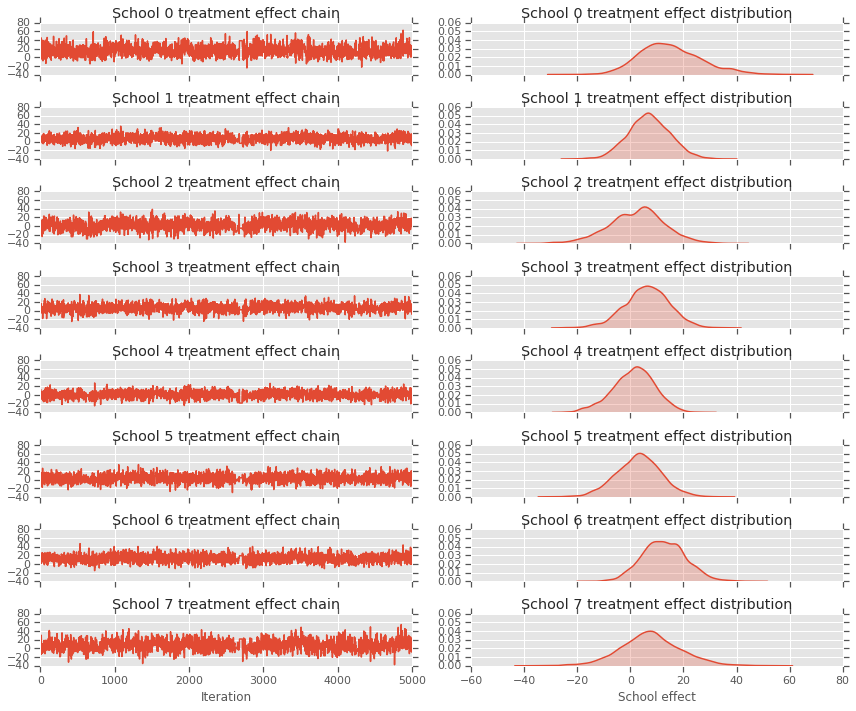
fig, axes = plt.subplots(8, 2, sharex='col', sharey='col')
fig.set_size_inches(12, 10)
for i in range(num_schools):
axes[i][0].plot(school_effects_samples[:,i].numpy())
axes[i][0].title.set_text("School {} treatment effect chain".format(i))
sns.kdeplot(school_effects_samples[:,i].numpy(), ax=axes[i][1], shade=True)
axes[i][1].title.set_text("School {} treatment effect distribution".format(i))
axes[num_schools - 1][0].set_xlabel("Iteration")
axes[num_schools - 1][1].set_xlabel("School effect")
fig.tight_layout()
plt.show()
print("E[avg_effect] = {}".format(np.mean(avg_effect)))
print("E[avg_stddev] = {}".format(np.mean(avg_stddev)))
print("E[school_effects_standard] =")
print(np.mean(school_effects_standard[:, ]))
print("E[school_effects] =")
print(np.mean(school_effects_samples[:, ], axis=0))
E[avg_effect] = 5.57183933258 E[avg_stddev] = 2.47738981247 E[school_effects_standard] = 0.08509017 E[school_effects] = [15.0051 7.103311 2.4552586 6.2744603 1.3364682 3.1125953 12.762501 7.743602 ]
# Compute the 95% interval for school_effects
school_effects_low = np.array([
np.percentile(school_effects_samples[:, i], 2.5) for i in range(num_schools)
])
school_effects_med = np.array([
np.percentile(school_effects_samples[:, i], 50) for i in range(num_schools)
])
school_effects_hi = np.array([
np.percentile(school_effects_samples[:, i], 97.5)
for i in range(num_schools)
])
fig, ax = plt.subplots(nrows=1, ncols=1, sharex=True)
ax.scatter(np.array(range(num_schools)), school_effects_med, color='red', s=60)
ax.scatter(
np.array(range(num_schools)) + 0.1, treatment_effects, color='blue', s=60)
plt.plot([-0.2, 7.4], [np.mean(avg_effect),
np.mean(avg_effect)], 'k', linestyle='--')
ax.errorbar(
np.array(range(8)),
school_effects_med,
yerr=[
school_effects_med - school_effects_low,
school_effects_hi - school_effects_med
],
fmt='none')
ax.legend(('avg_effect', 'HMC', 'Observed effect'), fontsize=14)
plt.xlabel('School')
plt.ylabel('Treatment effect')
plt.title('HMC estimated school treatment effects vs. observed data')
fig.set_size_inches(10, 8)
plt.show()
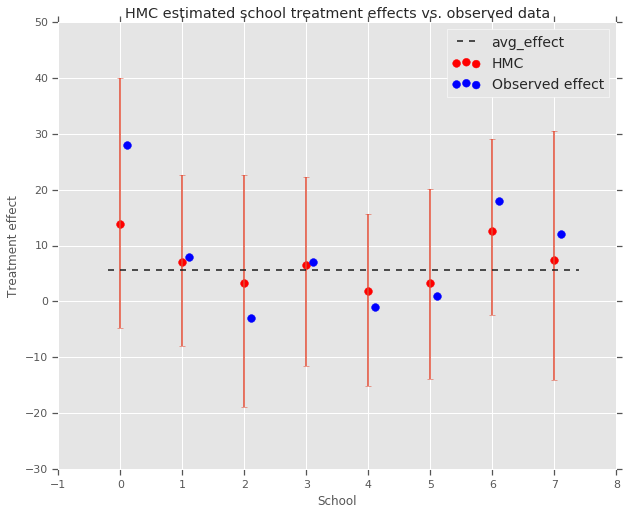
Biz grup doğru büzülme gözlemleyebilirsiniz avg_effect yukarıda.
print("Inferred posterior mean: {0:.2f}".format(
np.mean(school_effects_samples[:,])))
print("Inferred posterior mean se: {0:.2f}".format(
np.std(school_effects_samples[:,])))
Inferred posterior mean: 6.97 Inferred posterior mean se: 10.41
eleştiri
Yani arka öngörü dağılımı, yeni veri modeli almak için \(y^*\) gözlenen verilerin verilen \(y\):
\[ p(y^*|y) \propto \int_\theta p(y^* | \theta)p(\theta |y)d\theta\]
yeni veri oluşturmak için bu modelden arka dağılımı ve numunenin ortalama bunları ayarlamak için modelinde rastgele değişkenlerin değerleri geçersiz \(y^*\).
sample_shape = [5000]
_, _, _, predictive_treatment_effects = model.sample(
value=(tf.broadcast_to(np.mean(avg_effect, 0), sample_shape),
tf.broadcast_to(np.mean(avg_stddev, 0), sample_shape),
tf.broadcast_to(np.mean(school_effects_standard, 0),
sample_shape + [num_schools]),
None))
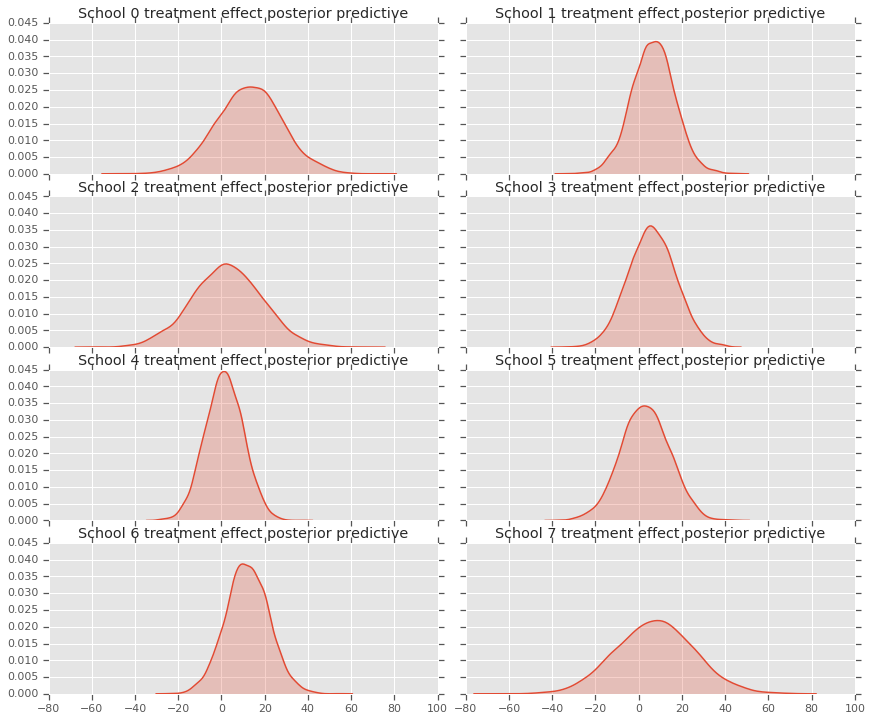
fig, axes = plt.subplots(4, 2, sharex=True, sharey=True)
fig.set_size_inches(12, 10)
fig.tight_layout()
for i, ax in enumerate(axes):
sns.kdeplot(predictive_treatment_effects[:, 2*i].numpy(),
ax=ax[0], shade=True)
ax[0].title.set_text(
"School {} treatment effect posterior predictive".format(2*i))
sns.kdeplot(predictive_treatment_effects[:, 2*i + 1].numpy(),
ax=ax[1], shade=True)
ax[1].title.set_text(
"School {} treatment effect posterior predictive".format(2*i + 1))
plt.show()
# The mean predicted treatment effects for each of the eight schools.
prediction = np.mean(predictive_treatment_effects, axis=0)
Tedavi etkileri verileri ile arka modelin tahminleri arasındaki artıklara bakabiliriz. Bunlar, nüfus ortalamasına doğru tahmini etkilerin küçülmesini gösteren yukarıdaki çizime karşılık gelir.
treatment_effects - prediction
array([14.905351 , 1.2838383, -5.6966295, 0.8327627, -2.3356671,
-2.0363257, 5.997898 , 4.3731265], dtype=float32)
Her okul için bir tahmin dağılımımız olduğu için artıkların dağılımını da düşünebiliriz.
residuals = treatment_effects - predictive_treatment_effects
fig, axes = plt.subplots(4, 2, sharex=True, sharey=True)
fig.set_size_inches(12, 10)
fig.tight_layout()
for i, ax in enumerate(axes):
sns.kdeplot(residuals[:, 2*i].numpy(), ax=ax[0], shade=True)
ax[0].title.set_text(
"School {} treatment effect residuals".format(2*i))
sns.kdeplot(residuals[:, 2*i + 1].numpy(), ax=ax[1], shade=True)
ax[1].title.set_text(
"School {} treatment effect residuals".format(2*i + 1))
plt.show()
Teşekkür
Bu eğitimde başlangıçta Edward 1,0 (yazılmış kaynak ). Bu sürümü yazmaya ve revize etmeye katkıda bulunan herkese teşekkür ederiz.
Referanslar
- Donald B. Rubin. Paralel randomize deneylerde tahmin. Eğitim İstatistikleri Dergisi, 6(4):377-401, 1981.
- Andrew Gelman, John Carlin, Hal Stern, David Dunson, Aki Vehtari ve Donald Rubin. Bayes Veri Analizi, Üçüncü Baskı. Chapman ve Hall/CRC, 2013.
