 Visualizza su TensorFlow.org Visualizza su TensorFlow.org |  Esegui in Google Colab Esegui in Google Colab |  Visualizza la fonte su GitHub Visualizza la fonte su GitHub |  Scarica il taccuino Scarica il taccuino |
Panoramica
Questo tutorial mostra come utilizzare tfio.image.decode_dicom_image in tensorflow IO per decodificare i file DICOM con tensorflow.
Configurazione e utilizzo
Scarica l'immagine DICOM
L'immagine DICOM utilizzato in questo tutorial è dal NIH torace X-ray set di dati .
Il NIH La radiografia del torace set di dati è costituito da 100.000 immagini de-identificati della radiografia del torace in formato PNG, forniti dal NIH Clinical Center e può essere scaricato tramite questo link .
Google Cloud offre anche una versione DICOM delle immagini, disponibile in Cloud Storage .
In questo tutorial, si scarica un file di esempio del set di dati dal repository GitHub
- Xiaosong Wang, Yifan Peng, Le Lu, Zhiyong Lu, Mohammadhadi Bagheri, Ronald Summers, ChestX-ray8: database dei raggi X del torace su scala ospedaliera e benchmark sulla classificazione e localizzazione debolmente supervisionate delle malattie del torace comuni, IEEE CVPR, pp. 3462 -3471, 2017
curl -OL https://github.com/tensorflow/io/raw/master/docs/tutorials/dicom/dicom_00000001_000.dcmls -l dicom_00000001_000.dcm
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 164 0 164 0 0 600 0 --:--:-- --:--:-- --:--:-- 598
100 1024k 100 1024k 0 0 1915k 0 --:--:-- --:--:-- --:--:-- 1915k
-rw-rw-r-- 1 kbuilder kokoro 1049332 Nov 22 03:47 dicom_00000001_000.dcm
Installa i pacchetti richiesti e riavvia il runtime
try:
# Use the Colab's preinstalled TensorFlow 2.x
%tensorflow_version 2.x
except:
pass
pip install tensorflow-io
Decodifica immagine DICOM
import matplotlib.pyplot as plt
import numpy as np
import tensorflow as tf
import tensorflow_io as tfio
image_bytes = tf.io.read_file('dicom_00000001_000.dcm')
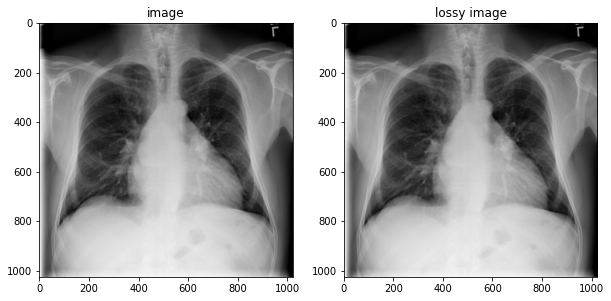
image = tfio.image.decode_dicom_image(image_bytes, dtype=tf.uint16)
skipped = tfio.image.decode_dicom_image(image_bytes, on_error='skip', dtype=tf.uint8)
lossy_image = tfio.image.decode_dicom_image(image_bytes, scale='auto', on_error='lossy', dtype=tf.uint8)
fig, axes = plt.subplots(1,2, figsize=(10,10))
axes[0].imshow(np.squeeze(image.numpy()), cmap='gray')
axes[0].set_title('image')
axes[1].imshow(np.squeeze(lossy_image.numpy()), cmap='gray')
axes[1].set_title('lossy image');
2021-11-22 03:47:53.016507: E tensorflow/stream_executor/cuda/cuda_driver.cc:271] failed call to cuInit: CUDA_ERROR_NO_DEVICE: no CUDA-capable device is detected
Decodifica i metadati DICOM e lavora con i tag
decode_dicom_data decodifica le informazioni dei tag. dicom_tags contiene informazioni utili come età e il sesso del paziente, in modo da poter usare DICOM tag come dicom_tags.PatientsAge e dicom_tags.PatientsSex . tensorflow_io prende in prestito la stessa notazione di tag dal pacchetto pydicom dicom.
tag_id = tfio.image.dicom_tags.PatientsAge
tag_value = tfio.image.decode_dicom_data(image_bytes,tag_id)
print(tag_value)
tf.Tensor(b'58', shape=(), dtype=string)
print(f"PatientsAge : {tag_value.numpy().decode('UTF-8')}")
PatientsAge : 58
tag_id = tfio.image.dicom_tags.PatientsSex
tag_value = tfio.image.decode_dicom_data(image_bytes,tag_id)
print(f"PatientsSex : {tag_value.numpy().decode('UTF-8')}")
PatientsSex : M
