 Посмотреть на TensorFlow.org Посмотреть на TensorFlow.org |  Запускаем в Google Colab Запускаем в Google Colab |  Посмотреть исходный код на GitHub Посмотреть исходный код на GitHub |  Скачать блокнот Скачать блокнот |
В этой коллаборации мы исследуем регрессию гауссовского процесса с помощью TensorFlow и TensorFlow Probability. Мы генерируем некоторые зашумленные наблюдения из некоторых известных функций и подгоняем модели GP к этим данным. Затем мы производим выборку из апостериорного GP и наносим на график значения выборочной функции по сеткам в их доменах.
Задний план
Пусть \(\mathcal{X}\) любое множество. Гауссовский процесс (ГП) представляет собой набор случайных величин , индексированных \(\mathcal{X}\) таким образом, что , если\(\{X_1, \ldots, X_n\} \subset \mathcal{X}\) любого конечное подмножество, предельная плотность\(p(X_1 = x_1, \ldots, X_n = x_n)\) является многомерным гауссовым. Любое гауссовское распределение полностью определяется его первым и вторым центральными моментами (средним и ковариантным), и GP не являются исключением. Мы можем указать GP полностью с точки зрения ее средней функции \(\mu : \mathcal{X} \to \mathbb{R}\) и ковариационной функции\(k : \mathcal{X} \times \mathcal{X} \to \mathbb{R}\). Большая часть выразительной силы GP заключена в выборе ковариационной функции. По разным причинам, корреляционная функция также называется функцией ядра. Требуется только , чтобы быть симметричной и положительно определенной (см , гл. 4 из Rasmussen & Williams ). Ниже мы используем ядро экспоненциальной квадратичной ковариации. Его форма
\[ k(x, x') := \sigma^2 \exp \left( \frac{\|x - x'\|^2}{\lambda^2} \right) \]
где \(\sigma^2\) называется «амплитуда» и \(\lambda\) длина шкалы. Параметры ядра можно выбрать с помощью процедуры оптимизации максимального правдоподобия.
Полный образец из GP содержит вещественную функцию по всему пространству\(\mathcal{X}\) и на практике нецелесообразно реализовать; часто выбирают набор точек для наблюдения за образцом и рисуют в этих точках значения функций. Это достигается путем выборки из подходящего (конечномерного) многомерного гаусса.
Обратите внимание, что согласно приведенному выше определению любое многомерное многомерное гауссовское распределение также является гауссовским процессом. Обычно, когда один относится к ГПУ, то подразумевается , что множество индексов некоторые \(\mathbb{R}^n\), и мы действительно будем делать это предположение здесь.
Распространенным применением гауссовских процессов в машинном обучении является регрессия гауссовских процессов. Идея заключается в том , что мы хотим оценить неизвестную функцию данные шумные наблюдений \(\{y_1, \ldots, y_N\}\) функции в конечном числе точек \(\{x_1, \ldots x_N\}.\) Мы представляем порождающий процесс
\[ \begin{align} f \sim \: & \textsf{GaussianProcess}\left( \text{mean_fn}=\mu(x), \text{covariance_fn}=k(x, x')\right) \\ y_i \sim \: & \textsf{Normal}\left( \text{loc}=f(x_i), \text{scale}=\sigma\right), i = 1, \ldots, N \end{align} \]
Как отмечалось выше, выборочную функцию невозможно вычислить, поскольку нам потребуется ее значение в бесконечном количестве точек. Вместо этого рассматривается конечная выборка из многомерного гауссовского.
\[ \begin{gather} \begin{bmatrix} f(x_1) \\ \vdots \\ f(x_N) \end{bmatrix} \sim \textsf{MultivariateNormal} \left( \: \text{loc}= \begin{bmatrix} \mu(x_1) \\ \vdots \\ \mu(x_N) \end{bmatrix} \:,\: \text{scale}= \begin{bmatrix} k(x_1, x_1) & \cdots & k(x_1, x_N) \\ \vdots & \ddots & \vdots \\ k(x_N, x_1) & \cdots & k(x_N, x_N) \\ \end{bmatrix}^{1/2} \: \right) \end{gather} \\ y_i \sim \textsf{Normal} \left( \text{loc}=f(x_i), \text{scale}=\sigma \right) \]
Обратите внимание на показатель степени \(\frac{1}{2}\) на ковариационной матрицы: это означает разложение Холецкого. Компенсация Холецкого необходима, потому что MVN - это семейное распределение в масштабе местоположения. К сожалению, разложение Холецкого вычислительно дорого, принимая \(O(N^3)\) времени и \(O(N^2)\) пространство. Большая часть литературы по GP сосредоточена на работе с этим, казалось бы, безобидным маленьким представителем.
Обычно априорную функцию среднего принимают постоянной, часто равной нулю. Кроме того, удобны некоторые условные обозначения. Один из часто пишет \(\mathbf{f}\) для конечного вектора выборочных значений функции. Ряд интересных обозначений используется для ковариационной матрицы в результате применения \(k\) к парам входов. После (Quiñonero-Кандела, 2005) , мы отмечаем , что компоненты матрицы ковариации значений функции при определенных входных точек. Таким образом , мы можем обозначить матрицу ковариации , как \(K_{AB}\), где \(A\) и \(B\) некоторые показатели сбора значений функции вдоль заданных размеров матрицы.
Например, с учетом наблюдаемых данных \((\mathbf{x}, \mathbf{y})\) с подразумеваемые значениями латентной функции \(\mathbf{f}\), мы можем написать
\[ K_{\mathbf{f},\mathbf{f} } = \begin{bmatrix} k(x_1, x_1) & \cdots & k(x_1, x_N) \\ \vdots & \ddots & \vdots \\ k(x_N, x_1) & \cdots & k(x_N, x_N) \\ \end{bmatrix} \]
Точно так же мы можем смешивать наборы входов, как в
\[ K_{\mathbf{f},*} = \begin{bmatrix} k(x_1, x^*_1) & \cdots & k(x_1, x^*_T) \\ \vdots & \ddots & \vdots \\ k(x_N, x^*_1) & \cdots & k(x_N, x^*_T) \\ \end{bmatrix} \]
где мы предположим , что есть \(N\) учебные входы и \(T\) входы тестов. Вышеупомянутый порождающий процесс может быть записан компактно как
\[ \begin{align} \mathbf{f} \sim \: & \textsf{MultivariateNormal} \left( \text{loc}=\mathbf{0}, \text{scale}=K_{\mathbf{f},\mathbf{f} }^{1/2} \right) \\ y_i \sim \: & \textsf{Normal} \left( \text{loc}=f_i, \text{scale}=\sigma \right), i = 1, \ldots, N \end{align} \]
Операции выборки в первой строке дает конечное множество \(N\) значений функции от многомерного гауссова - не целая функцию как в приведенном выше GP рисовать обозначения. Вторая строка описывает набор \(N\) извлекает из одномерных гауссиан с центром в различных значениях функции, с фиксированным наблюдением шумом \(\sigma^2\).
Имея указанную выше генеративную модель, мы можем перейти к рассмотрению проблемы апостериорного вывода. Это дает апостериорное распределение по значениям функции в новом наборе контрольных точек, обусловленное наблюдаемыми зашумленными данными из описанного выше процесса.
С учетом указанными выше обозначениями на месте, мы можем компактно записать заднее прогностическое распределение по будущему (шумовые) наблюдения обусловливающих соответствующих входов и подготовки данных следующим образом (для получения более подробной информации см §2.2 из Rasmussen & Williams ).
\[ \mathbf{y}^* \mid \mathbf{x}^*, \mathbf{x}, \mathbf{y} \sim \textsf{Normal} \left( \text{loc}=\mathbf{\mu}^*, \text{scale}=(\Sigma^*)^{1/2} \right), \]
где
\[ \mathbf{\mu}^* = K_{*,\mathbf{f} }\left(K_{\mathbf{f},\mathbf{f} } + \sigma^2 I \right)^{-1} \mathbf{y} \]
и
\[ \Sigma^* = K_{*,*} - K_{*,\mathbf{f} } \left(K_{\mathbf{f},\mathbf{f} } + \sigma^2 I \right)^{-1} K_{\mathbf{f},*} \]
Импорт
import time
import numpy as np
import matplotlib.pyplot as plt
import tensorflow.compat.v2 as tf
import tensorflow_probability as tfp
tfb = tfp.bijectors
tfd = tfp.distributions
tfk = tfp.math.psd_kernels
tf.enable_v2_behavior()
from mpl_toolkits.mplot3d import Axes3D
%pylab inline
# Configure plot defaults
plt.rcParams['axes.facecolor'] = 'white'
plt.rcParams['grid.color'] = '#666666'
%config InlineBackend.figure_format = 'png'
Populating the interactive namespace from numpy and matplotlib
Пример: точная регрессия GP на зашумленных синусоидальных данных
Здесь мы генерируем данные обучения из зашумленной синусоиды, а затем выбираем набор кривых из апостериорной модели регрессии GP. Мы используем Адам оптимизировать гиперпараметры ядра (мы сводим к минимуму отрицательного логарифма вероятности данных по настоятелю). Мы строим тренировочную кривую, затем истинную функцию и апостериорные образцы.
def sinusoid(x):
return np.sin(3 * np.pi * x[..., 0])
def generate_1d_data(num_training_points, observation_noise_variance):
"""Generate noisy sinusoidal observations at a random set of points.
Returns:
observation_index_points, observations
"""
index_points_ = np.random.uniform(-1., 1., (num_training_points, 1))
index_points_ = index_points_.astype(np.float64)
# y = f(x) + noise
observations_ = (sinusoid(index_points_) +
np.random.normal(loc=0,
scale=np.sqrt(observation_noise_variance),
size=(num_training_points)))
return index_points_, observations_
# Generate training data with a known noise level (we'll later try to recover
# this value from the data).
NUM_TRAINING_POINTS = 100
observation_index_points_, observations_ = generate_1d_data(
num_training_points=NUM_TRAINING_POINTS,
observation_noise_variance=.1)
Мы поместим априорные на гиперпараметрах ядра и записать совместное распределение гиперпараметров и наблюдаемых данных с использованием tfd.JointDistributionNamed .
def build_gp(amplitude, length_scale, observation_noise_variance):
"""Defines the conditional dist. of GP outputs, given kernel parameters."""
# Create the covariance kernel, which will be shared between the prior (which we
# use for maximum likelihood training) and the posterior (which we use for
# posterior predictive sampling)
kernel = tfk.ExponentiatedQuadratic(amplitude, length_scale)
# Create the GP prior distribution, which we will use to train the model
# parameters.
return tfd.GaussianProcess(
kernel=kernel,
index_points=observation_index_points_,
observation_noise_variance=observation_noise_variance)
gp_joint_model = tfd.JointDistributionNamed({
'amplitude': tfd.LogNormal(loc=0., scale=np.float64(1.)),
'length_scale': tfd.LogNormal(loc=0., scale=np.float64(1.)),
'observation_noise_variance': tfd.LogNormal(loc=0., scale=np.float64(1.)),
'observations': build_gp,
})
Мы можем проверить работоспособность нашей реализации, убедившись, что мы можем производить выборку из предыдущего, и вычислить логарифмическую плотность выборки.
x = gp_joint_model.sample()
lp = gp_joint_model.log_prob(x)
print("sampled {}".format(x))
print("log_prob of sample: {}".format(lp))
sampled {'observation_noise_variance': <tf.Tensor: shape=(), dtype=float64, numpy=2.067952217184325>, 'length_scale': <tf.Tensor: shape=(), dtype=float64, numpy=1.154435715487831>, 'amplitude': <tf.Tensor: shape=(), dtype=float64, numpy=5.383850737703549>, 'observations': <tf.Tensor: shape=(100,), dtype=float64, numpy=
array([-2.37070577, -2.05363838, -0.95152824, 3.73509388, -0.2912646 ,
0.46112342, -1.98018513, -2.10295857, -1.33589756, -2.23027226,
-2.25081374, -0.89450835, -2.54196452, 1.46621647, 2.32016193,
5.82399989, 2.27241034, -0.67523432, -1.89150197, -1.39834474,
-2.33954116, 0.7785609 , -1.42763627, -0.57389025, -0.18226098,
-3.45098732, 0.27986652, -3.64532398, -1.28635204, -2.42362875,
0.01107288, -2.53222176, -2.0886136 , -5.54047694, -2.18389607,
-1.11665628, -3.07161217, -2.06070336, -0.84464262, 1.29238438,
-0.64973999, -2.63805504, -3.93317576, 0.65546645, 2.24721181,
-0.73403676, 5.31628298, -1.2208384 , 4.77782252, -1.42978168,
-3.3089274 , 3.25370494, 3.02117591, -1.54862932, -1.07360811,
1.2004856 , -4.3017773 , -4.95787789, -1.95245901, -2.15960839,
-3.78592731, -1.74096185, 3.54891595, 0.56294143, 1.15288455,
-0.77323696, 2.34430694, -1.05302007, -0.7514684 , -0.98321063,
-3.01300144, -3.00033274, 0.44200837, 0.45060886, -1.84497318,
-1.89616746, -2.15647664, -2.65672581, -3.65493379, 1.70923375,
-3.88695218, -0.05151283, 4.51906677, -2.28117003, 3.03032793,
-1.47713194, -0.35625273, 3.73501587, -2.09328047, -0.60665614,
-0.78177188, -0.67298545, 2.97436033, -0.29407932, 2.98482427,
-1.54951178, 2.79206821, 4.2225733 , 2.56265198, 2.80373284])>}
log_prob of sample: -194.96442183797524
Теперь давайте оптимизируем, чтобы найти значения параметров с наибольшей апостериорной вероятностью. Мы определим переменную для каждого параметра и ограничим их значения положительными.
# Create the trainable model parameters, which we'll subsequently optimize.
# Note that we constrain them to be strictly positive.
constrain_positive = tfb.Shift(np.finfo(np.float64).tiny)(tfb.Exp())
amplitude_var = tfp.util.TransformedVariable(
initial_value=1.,
bijector=constrain_positive,
name='amplitude',
dtype=np.float64)
length_scale_var = tfp.util.TransformedVariable(
initial_value=1.,
bijector=constrain_positive,
name='length_scale',
dtype=np.float64)
observation_noise_variance_var = tfp.util.TransformedVariable(
initial_value=1.,
bijector=constrain_positive,
name='observation_noise_variance_var',
dtype=np.float64)
trainable_variables = [v.trainable_variables[0] for v in
[amplitude_var,
length_scale_var,
observation_noise_variance_var]]
Для проветривания модели на наших данных наблюдений, мы определим target_log_prob функцию, которая принимает (все еще должны быть выведено) ядра гиперпараметров.
def target_log_prob(amplitude, length_scale, observation_noise_variance):
return gp_joint_model.log_prob({
'amplitude': amplitude,
'length_scale': length_scale,
'observation_noise_variance': observation_noise_variance,
'observations': observations_
})
# Now we optimize the model parameters.
num_iters = 1000
optimizer = tf.optimizers.Adam(learning_rate=.01)
# Use `tf.function` to trace the loss for more efficient evaluation.
@tf.function(autograph=False, jit_compile=False)
def train_model():
with tf.GradientTape() as tape:
loss = -target_log_prob(amplitude_var, length_scale_var,
observation_noise_variance_var)
grads = tape.gradient(loss, trainable_variables)
optimizer.apply_gradients(zip(grads, trainable_variables))
return loss
# Store the likelihood values during training, so we can plot the progress
lls_ = np.zeros(num_iters, np.float64)
for i in range(num_iters):
loss = train_model()
lls_[i] = loss
print('Trained parameters:')
print('amplitude: {}'.format(amplitude_var._value().numpy()))
print('length_scale: {}'.format(length_scale_var._value().numpy()))
print('observation_noise_variance: {}'.format(observation_noise_variance_var._value().numpy()))
Trained parameters: amplitude: 0.9176153445125278 length_scale: 0.18444082442910079 observation_noise_variance: 0.0880273312850989
# Plot the loss evolution
plt.figure(figsize=(12, 4))
plt.plot(lls_)
plt.xlabel("Training iteration")
plt.ylabel("Log marginal likelihood")
plt.show()

# Having trained the model, we'd like to sample from the posterior conditioned
# on observations. We'd like the samples to be at points other than the training
# inputs.
predictive_index_points_ = np.linspace(-1.2, 1.2, 200, dtype=np.float64)
# Reshape to [200, 1] -- 1 is the dimensionality of the feature space.
predictive_index_points_ = predictive_index_points_[..., np.newaxis]
optimized_kernel = tfk.ExponentiatedQuadratic(amplitude_var, length_scale_var)
gprm = tfd.GaussianProcessRegressionModel(
kernel=optimized_kernel,
index_points=predictive_index_points_,
observation_index_points=observation_index_points_,
observations=observations_,
observation_noise_variance=observation_noise_variance_var,
predictive_noise_variance=0.)
# Create op to draw 50 independent samples, each of which is a *joint* draw
# from the posterior at the predictive_index_points_. Since we have 200 input
# locations as defined above, this posterior distribution over corresponding
# function values is a 200-dimensional multivariate Gaussian distribution!
num_samples = 50
samples = gprm.sample(num_samples)
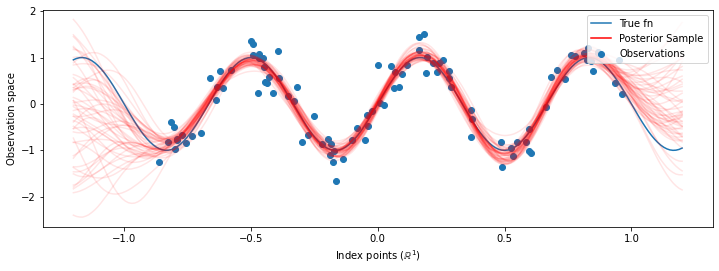
# Plot the true function, observations, and posterior samples.
plt.figure(figsize=(12, 4))
plt.plot(predictive_index_points_, sinusoid(predictive_index_points_),
label='True fn')
plt.scatter(observation_index_points_[:, 0], observations_,
label='Observations')
for i in range(num_samples):
plt.plot(predictive_index_points_, samples[i, :], c='r', alpha=.1,
label='Posterior Sample' if i == 0 else None)
leg = plt.legend(loc='upper right')
for lh in leg.legendHandles:
lh.set_alpha(1)
plt.xlabel(r"Index points ($\mathbb{R}^1$)")
plt.ylabel("Observation space")
plt.show()
Маргинализация гиперпараметров с помощью HMC
Вместо оптимизации гиперпараметров давайте попробуем интегрировать их с гамильтонианом Монте-Карло. Сначала мы определим и запустим семплер, чтобы приблизительно извлечь из апостериорного распределения гиперпараметры ядра с учетом наблюдений.
num_results = 100
num_burnin_steps = 50
sampler = tfp.mcmc.TransformedTransitionKernel(
tfp.mcmc.NoUTurnSampler(
target_log_prob_fn=target_log_prob,
step_size=tf.cast(0.1, tf.float64)),
bijector=[constrain_positive, constrain_positive, constrain_positive])
adaptive_sampler = tfp.mcmc.DualAveragingStepSizeAdaptation(
inner_kernel=sampler,
num_adaptation_steps=int(0.8 * num_burnin_steps),
target_accept_prob=tf.cast(0.75, tf.float64))
initial_state = [tf.cast(x, tf.float64) for x in [1., 1., 1.]]
# Speed up sampling by tracing with `tf.function`.
@tf.function(autograph=False, jit_compile=False)
def do_sampling():
return tfp.mcmc.sample_chain(
kernel=adaptive_sampler,
current_state=initial_state,
num_results=num_results,
num_burnin_steps=num_burnin_steps,
trace_fn=lambda current_state, kernel_results: kernel_results)
t0 = time.time()
samples, kernel_results = do_sampling()
t1 = time.time()
print("Inference ran in {:.2f}s.".format(t1-t0))
Inference ran in 9.00s.
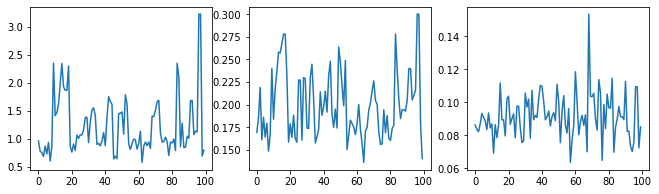
Давайте проверим работоспособность семплера, исследуя следы гиперпараметров.
(amplitude_samples,
length_scale_samples,
observation_noise_variance_samples) = samples
f = plt.figure(figsize=[15, 3])
for i, s in enumerate(samples):
ax = f.add_subplot(1, len(samples) + 1, i + 1)
ax.plot(s)
Теперь вместо того , чтобы строить один GP с оптимизированными гиперпараметрами, мы строим заднее прогностическое распределение в виде смеси ВОПА, каждый из которых определяется образцом от заднего распределения по гиперпараметрам. Это приблизительно объединяет апостериорные параметры с помощью выборки Монте-Карло для вычисления предельного прогнозируемого распределения в ненаблюдаемых местах.
# The sampled hyperparams have a leading batch dimension, `[num_results, ...]`,
# so they construct a *batch* of kernels.
batch_of_posterior_kernels = tfk.ExponentiatedQuadratic(
amplitude_samples, length_scale_samples)
# The batch of kernels creates a batch of GP predictive models, one for each
# posterior sample.
batch_gprm = tfd.GaussianProcessRegressionModel(
kernel=batch_of_posterior_kernels,
index_points=predictive_index_points_,
observation_index_points=observation_index_points_,
observations=observations_,
observation_noise_variance=observation_noise_variance_samples,
predictive_noise_variance=0.)
# To construct the marginal predictive distribution, we average with uniform
# weight over the posterior samples.
predictive_gprm = tfd.MixtureSameFamily(
mixture_distribution=tfd.Categorical(logits=tf.zeros([num_results])),
components_distribution=batch_gprm)
num_samples = 50
samples = predictive_gprm.sample(num_samples)
# Plot the true function, observations, and posterior samples.
plt.figure(figsize=(12, 4))
plt.plot(predictive_index_points_, sinusoid(predictive_index_points_),
label='True fn')
plt.scatter(observation_index_points_[:, 0], observations_,
label='Observations')
for i in range(num_samples):
plt.plot(predictive_index_points_, samples[i, :], c='r', alpha=.1,
label='Posterior Sample' if i == 0 else None)
leg = plt.legend(loc='upper right')
for lh in leg.legendHandles:
lh.set_alpha(1)
plt.xlabel(r"Index points ($\mathbb{R}^1$)")
plt.ylabel("Observation space")
plt.show()
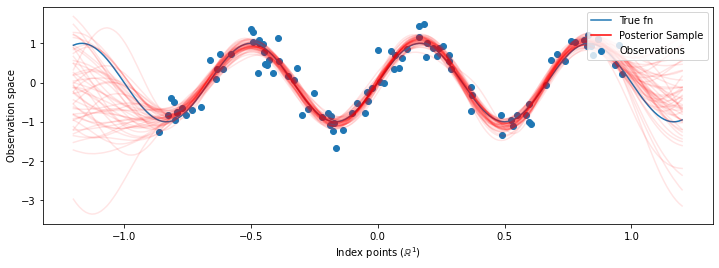
Хотя различия в этом случае незначительны, в целом мы ожидаем, что апостериорное прогнозирующее распределение будет лучше обобщать (давать более высокую вероятность удерживаемым данным), чем просто использовать наиболее вероятные параметры, как мы делали выше.
