 Lihat di TensorFlow.org Lihat di TensorFlow.org |  Jalankan di Google Colab Jalankan di Google Colab |  Lihat sumber di GitHub Lihat sumber di GitHub |  Unduh buku catatan Unduh buku catatan |
Dalam colab ini, kami mengeksplorasi regresi proses Gaussian menggunakan TensorFlow dan TensorFlow Probability. Kami menghasilkan beberapa pengamatan bising dari beberapa fungsi yang diketahui dan menyesuaikan model GP dengan data tersebut. Kami kemudian mengambil sampel dari GP posterior dan memplot nilai fungsi sampel di atas grid di domain mereka.
Latar belakang
Mari \(\mathcal{X}\) menjadi setiap set. Sebuah proses Gaussian (GP) adalah kumpulan dari variabel acak diindeks oleh \(\mathcal{X}\) sehingga jika\(\{X_1, \ldots, X_n\} \subset \mathcal{X}\) adalah subset terbatas, marjinal kepadatan\(p(X_1 = x_1, \ldots, X_n = x_n)\) adalah multivariat Gaussian. Setiap distribusi Gaussian sepenuhnya ditentukan oleh momen pusat pertama dan kedua (rata-rata dan kovarians), dan GP tidak terkecuali. Kita dapat menentukan GP sepenuhnya dalam hal rata-rata fungsinya \(\mu : \mathcal{X} \to \mathbb{R}\) dan kovarians fungsi\(k : \mathcal{X} \times \mathcal{X} \to \mathbb{R}\). Sebagian besar kekuatan ekspresif GP dikemas dalam pilihan fungsi kovarians. Karena berbagai alasan, fungsi kovarians juga disebut sebagai fungsi kernel. Hal ini diperlukan hanya untuk simetris dan positif-yang pasti (lihat Ch. 4 dari Rasmussen & Williams ). Di bawah ini kami menggunakan kernel kovarians ExpontiatedQuadratic. Bentuknya adalah
\[ k(x, x') := \sigma^2 \exp \left( \frac{\|x - x'\|^2}{\lambda^2} \right) \]
di mana \(\sigma^2\) disebut 'amplitudo' dan \(\lambda\) skala panjang. Parameter kernel dapat dipilih melalui prosedur optimisasi kemungkinan maksimum.
Sebuah sampel penuh dari GP terdiri dari fungsi bernilai real di seluruh ruang\(\mathcal{X}\) dan dalam praktek tidak praktis untuk mewujudkan; seringkali seseorang memilih sekumpulan titik untuk mengamati sampel dan menggambar nilai fungsi pada titik-titik ini. Hal ini dicapai dengan pengambilan sampel dari Gaussian multivariat (dimensi terbatas) yang sesuai.
Perhatikan bahwa, menurut definisi di atas, setiap distribusi Gaussian multivariat berdimensi-hingga juga merupakan proses Gaussian. Biasanya, ketika salah satu mengacu pada GP, itu tersirat bahwa indeks set beberapa \(\mathbb{R}^n\)dan kami memang akan membuat asumsi ini di sini.
Aplikasi umum proses Gaussian dalam pembelajaran mesin adalah regresi proses Gaussian. Idenya adalah bahwa kita ingin memperkirakan fungsi yang tidak diketahui pengamatan berisik diberikan \(\{y_1, \ldots, y_N\}\) fungsi di sejumlah terbatas poin \(\{x_1, \ldots x_N\}.\) Kami membayangkan proses generatif
\[ \begin{align} f \sim \: & \textsf{GaussianProcess}\left( \text{mean_fn}=\mu(x), \text{covariance_fn}=k(x, x')\right) \\ y_i \sim \: & \textsf{Normal}\left( \text{loc}=f(x_i), \text{scale}=\sigma\right), i = 1, \ldots, N \end{align} \]
Seperti disebutkan di atas, fungsi sampel tidak mungkin dihitung, karena kita akan membutuhkan nilainya pada jumlah titik yang tak terbatas. Sebaliknya, kita menganggap sampel terbatas dari Gaussian multivariat.
\[ \begin{gather} \begin{bmatrix} f(x_1) \\ \vdots \\ f(x_N) \end{bmatrix} \sim \textsf{MultivariateNormal} \left( \: \text{loc}= \begin{bmatrix} \mu(x_1) \\ \vdots \\ \mu(x_N) \end{bmatrix} \:,\: \text{scale}= \begin{bmatrix} k(x_1, x_1) & \cdots & k(x_1, x_N) \\ \vdots & \ddots & \vdots \\ k(x_N, x_1) & \cdots & k(x_N, x_N) \\ \end{bmatrix}^{1/2} \: \right) \end{gather} \\ y_i \sim \textsf{Normal} \left( \text{loc}=f(x_i), \text{scale}=\sigma \right) \]
Catatan eksponen \(\frac{1}{2}\) pada matriks kovarians: ini menunjukkan dekomposisi Cholesky. Menghitung Cholesky diperlukan karena MVN adalah distribusi keluarga skala lokasi. Sayangnya dekomposisi Cholesky adalah komputasi mahal, mengambil \(O(N^3)\) waktu dan \(O(N^2)\) ruang. Sebagian besar literatur GP difokuskan pada penanganan eksponen kecil yang tampaknya tidak berbahaya ini.
Adalah umum untuk mengambil fungsi rata-rata sebelumnya konstan, seringkali nol. Juga, beberapa konvensi notasi nyaman. Satu sering menulis \(\mathbf{f}\) untuk vektor terbatas nilai-nilai fungsi sampel. Sejumlah notasi yang menarik digunakan untuk matriks kovarians yang dihasilkan dari penerapan \(k\) untuk pasang masukan. Berikut (Quiñonero-Candela, 2005) , kami mencatat bahwa komponen matriks yang covariances nilai fungsi pada titik-titik masukan tertentu. Dengan demikian kita dapat menunjukkan matriks kovarians sebagai \(K_{AB}\)mana \(A\) dan \(B\) adalah beberapa indikator koleksi nilai fungsi sepanjang dimensi matriks yang diberikan.
Sebagai contoh, mengingat data yang diamati \((\mathbf{x}, \mathbf{y})\) dengan nilai-nilai fungsi laten tersirat \(\mathbf{f}\), kita dapat menulis
\[ K_{\mathbf{f},\mathbf{f} } = \begin{bmatrix} k(x_1, x_1) & \cdots & k(x_1, x_N) \\ \vdots & \ddots & \vdots \\ k(x_N, x_1) & \cdots & k(x_N, x_N) \\ \end{bmatrix} \]
Demikian pula, kita dapat mencampur set input, seperti pada
\[ K_{\mathbf{f},*} = \begin{bmatrix} k(x_1, x^*_1) & \cdots & k(x_1, x^*_T) \\ \vdots & \ddots & \vdots \\ k(x_N, x^*_1) & \cdots & k(x_N, x^*_T) \\ \end{bmatrix} \]
di mana kita kira ada \(N\) pelatihan masukan, dan \(T\) tes input. Proses generatif di atas kemudian dapat ditulis secara ringkas sebagai
\[ \begin{align} \mathbf{f} \sim \: & \textsf{MultivariateNormal} \left( \text{loc}=\mathbf{0}, \text{scale}=K_{\mathbf{f},\mathbf{f} }^{1/2} \right) \\ y_i \sim \: & \textsf{Normal} \left( \text{loc}=f_i, \text{scale}=\sigma \right), i = 1, \ldots, N \end{align} \]
Operasi pengambilan sampel di baris pertama menghasilkan satu set terbatas \(N\) nilai fungsi dari Gaussian multivariat - tidak seluruh fungsi seperti di atas GP menarik notasi. Baris kedua menggambarkan koleksi \(N\) menarik dari univariat Gaussians berpusat di berbagai nilai fungsi, dengan tetap pengamatan noise \(\sigma^2\).
Dengan model generatif di atas, kita dapat melanjutkan untuk mempertimbangkan masalah inferensi posterior. Ini menghasilkan distribusi posterior atas nilai fungsi pada set titik uji baru, yang dikondisikan pada data noise yang diamati dari proses di atas.
Dengan notasi di atas di tempat, kita kompak dapat menulis distribusi prediksi posterior atas masa depan (berisik) pengamatan tergantung pada sesuai masukan dan data pelatihan sebagai berikut (untuk lebih jelasnya, lihat §2.2 dari Rasmussen & Williams ).
\[ \mathbf{y}^* \mid \mathbf{x}^*, \mathbf{x}, \mathbf{y} \sim \textsf{Normal} \left( \text{loc}=\mathbf{\mu}^*, \text{scale}=(\Sigma^*)^{1/2} \right), \]
di mana
\[ \mathbf{\mu}^* = K_{*,\mathbf{f} }\left(K_{\mathbf{f},\mathbf{f} } + \sigma^2 I \right)^{-1} \mathbf{y} \]
dan
\[ \Sigma^* = K_{*,*} - K_{*,\mathbf{f} } \left(K_{\mathbf{f},\mathbf{f} } + \sigma^2 I \right)^{-1} K_{\mathbf{f},*} \]
Impor
import time
import numpy as np
import matplotlib.pyplot as plt
import tensorflow.compat.v2 as tf
import tensorflow_probability as tfp
tfb = tfp.bijectors
tfd = tfp.distributions
tfk = tfp.math.psd_kernels
tf.enable_v2_behavior()
from mpl_toolkits.mplot3d import Axes3D
%pylab inline
# Configure plot defaults
plt.rcParams['axes.facecolor'] = 'white'
plt.rcParams['grid.color'] = '#666666'
%config InlineBackend.figure_format = 'png'
Populating the interactive namespace from numpy and matplotlib
Contoh: Regresi GP Tepat pada Data Sinusoidal Bising
Di sini kami menghasilkan data pelatihan dari sinusoid yang bising, kemudian mengambil sampel sekelompok kurva dari posterior model regresi GP. Kami menggunakan Adam untuk mengoptimalkan hyperparameters kernel (kita meminimalkan log kemungkinan negatif dari data di bawah sebelumnya). Kami memplot kurva pelatihan, diikuti oleh fungsi sebenarnya dan sampel posterior.
def sinusoid(x):
return np.sin(3 * np.pi * x[..., 0])
def generate_1d_data(num_training_points, observation_noise_variance):
"""Generate noisy sinusoidal observations at a random set of points.
Returns:
observation_index_points, observations
"""
index_points_ = np.random.uniform(-1., 1., (num_training_points, 1))
index_points_ = index_points_.astype(np.float64)
# y = f(x) + noise
observations_ = (sinusoid(index_points_) +
np.random.normal(loc=0,
scale=np.sqrt(observation_noise_variance),
size=(num_training_points)))
return index_points_, observations_
# Generate training data with a known noise level (we'll later try to recover
# this value from the data).
NUM_TRAINING_POINTS = 100
observation_index_points_, observations_ = generate_1d_data(
num_training_points=NUM_TRAINING_POINTS,
observation_noise_variance=.1)
Kami akan menempatkan prior pada hyperparameters kernel, dan menulis distribusi bersama dari hyperparameters dan data yang diamati menggunakan tfd.JointDistributionNamed .
def build_gp(amplitude, length_scale, observation_noise_variance):
"""Defines the conditional dist. of GP outputs, given kernel parameters."""
# Create the covariance kernel, which will be shared between the prior (which we
# use for maximum likelihood training) and the posterior (which we use for
# posterior predictive sampling)
kernel = tfk.ExponentiatedQuadratic(amplitude, length_scale)
# Create the GP prior distribution, which we will use to train the model
# parameters.
return tfd.GaussianProcess(
kernel=kernel,
index_points=observation_index_points_,
observation_noise_variance=observation_noise_variance)
gp_joint_model = tfd.JointDistributionNamed({
'amplitude': tfd.LogNormal(loc=0., scale=np.float64(1.)),
'length_scale': tfd.LogNormal(loc=0., scale=np.float64(1.)),
'observation_noise_variance': tfd.LogNormal(loc=0., scale=np.float64(1.)),
'observations': build_gp,
})
Kami dapat memeriksa kewarasan implementasi kami dengan memverifikasi bahwa kami dapat mengambil sampel dari sebelumnya, dan menghitung kepadatan log dari sampel.
x = gp_joint_model.sample()
lp = gp_joint_model.log_prob(x)
print("sampled {}".format(x))
print("log_prob of sample: {}".format(lp))
sampled {'observation_noise_variance': <tf.Tensor: shape=(), dtype=float64, numpy=2.067952217184325>, 'length_scale': <tf.Tensor: shape=(), dtype=float64, numpy=1.154435715487831>, 'amplitude': <tf.Tensor: shape=(), dtype=float64, numpy=5.383850737703549>, 'observations': <tf.Tensor: shape=(100,), dtype=float64, numpy=
array([-2.37070577, -2.05363838, -0.95152824, 3.73509388, -0.2912646 ,
0.46112342, -1.98018513, -2.10295857, -1.33589756, -2.23027226,
-2.25081374, -0.89450835, -2.54196452, 1.46621647, 2.32016193,
5.82399989, 2.27241034, -0.67523432, -1.89150197, -1.39834474,
-2.33954116, 0.7785609 , -1.42763627, -0.57389025, -0.18226098,
-3.45098732, 0.27986652, -3.64532398, -1.28635204, -2.42362875,
0.01107288, -2.53222176, -2.0886136 , -5.54047694, -2.18389607,
-1.11665628, -3.07161217, -2.06070336, -0.84464262, 1.29238438,
-0.64973999, -2.63805504, -3.93317576, 0.65546645, 2.24721181,
-0.73403676, 5.31628298, -1.2208384 , 4.77782252, -1.42978168,
-3.3089274 , 3.25370494, 3.02117591, -1.54862932, -1.07360811,
1.2004856 , -4.3017773 , -4.95787789, -1.95245901, -2.15960839,
-3.78592731, -1.74096185, 3.54891595, 0.56294143, 1.15288455,
-0.77323696, 2.34430694, -1.05302007, -0.7514684 , -0.98321063,
-3.01300144, -3.00033274, 0.44200837, 0.45060886, -1.84497318,
-1.89616746, -2.15647664, -2.65672581, -3.65493379, 1.70923375,
-3.88695218, -0.05151283, 4.51906677, -2.28117003, 3.03032793,
-1.47713194, -0.35625273, 3.73501587, -2.09328047, -0.60665614,
-0.78177188, -0.67298545, 2.97436033, -0.29407932, 2.98482427,
-1.54951178, 2.79206821, 4.2225733 , 2.56265198, 2.80373284])>}
log_prob of sample: -194.96442183797524
Sekarang mari kita optimalkan untuk menemukan nilai parameter dengan probabilitas posterior tertinggi. Kami akan mendefinisikan variabel untuk setiap parameter, dan membatasi nilainya menjadi positif.
# Create the trainable model parameters, which we'll subsequently optimize.
# Note that we constrain them to be strictly positive.
constrain_positive = tfb.Shift(np.finfo(np.float64).tiny)(tfb.Exp())
amplitude_var = tfp.util.TransformedVariable(
initial_value=1.,
bijector=constrain_positive,
name='amplitude',
dtype=np.float64)
length_scale_var = tfp.util.TransformedVariable(
initial_value=1.,
bijector=constrain_positive,
name='length_scale',
dtype=np.float64)
observation_noise_variance_var = tfp.util.TransformedVariable(
initial_value=1.,
bijector=constrain_positive,
name='observation_noise_variance_var',
dtype=np.float64)
trainable_variables = [v.trainable_variables[0] for v in
[amplitude_var,
length_scale_var,
observation_noise_variance_var]]
Untuk kondisi model pada data yang diamati kami, kami akan menentukan target_log_prob fungsi, yang mengambil (masih harus disimpulkan) hyperparameters kernel.
def target_log_prob(amplitude, length_scale, observation_noise_variance):
return gp_joint_model.log_prob({
'amplitude': amplitude,
'length_scale': length_scale,
'observation_noise_variance': observation_noise_variance,
'observations': observations_
})
# Now we optimize the model parameters.
num_iters = 1000
optimizer = tf.optimizers.Adam(learning_rate=.01)
# Use `tf.function` to trace the loss for more efficient evaluation.
@tf.function(autograph=False, jit_compile=False)
def train_model():
with tf.GradientTape() as tape:
loss = -target_log_prob(amplitude_var, length_scale_var,
observation_noise_variance_var)
grads = tape.gradient(loss, trainable_variables)
optimizer.apply_gradients(zip(grads, trainable_variables))
return loss
# Store the likelihood values during training, so we can plot the progress
lls_ = np.zeros(num_iters, np.float64)
for i in range(num_iters):
loss = train_model()
lls_[i] = loss
print('Trained parameters:')
print('amplitude: {}'.format(amplitude_var._value().numpy()))
print('length_scale: {}'.format(length_scale_var._value().numpy()))
print('observation_noise_variance: {}'.format(observation_noise_variance_var._value().numpy()))
Trained parameters: amplitude: 0.9176153445125278 length_scale: 0.18444082442910079 observation_noise_variance: 0.0880273312850989
# Plot the loss evolution
plt.figure(figsize=(12, 4))
plt.plot(lls_)
plt.xlabel("Training iteration")
plt.ylabel("Log marginal likelihood")
plt.show()

# Having trained the model, we'd like to sample from the posterior conditioned
# on observations. We'd like the samples to be at points other than the training
# inputs.
predictive_index_points_ = np.linspace(-1.2, 1.2, 200, dtype=np.float64)
# Reshape to [200, 1] -- 1 is the dimensionality of the feature space.
predictive_index_points_ = predictive_index_points_[..., np.newaxis]
optimized_kernel = tfk.ExponentiatedQuadratic(amplitude_var, length_scale_var)
gprm = tfd.GaussianProcessRegressionModel(
kernel=optimized_kernel,
index_points=predictive_index_points_,
observation_index_points=observation_index_points_,
observations=observations_,
observation_noise_variance=observation_noise_variance_var,
predictive_noise_variance=0.)
# Create op to draw 50 independent samples, each of which is a *joint* draw
# from the posterior at the predictive_index_points_. Since we have 200 input
# locations as defined above, this posterior distribution over corresponding
# function values is a 200-dimensional multivariate Gaussian distribution!
num_samples = 50
samples = gprm.sample(num_samples)
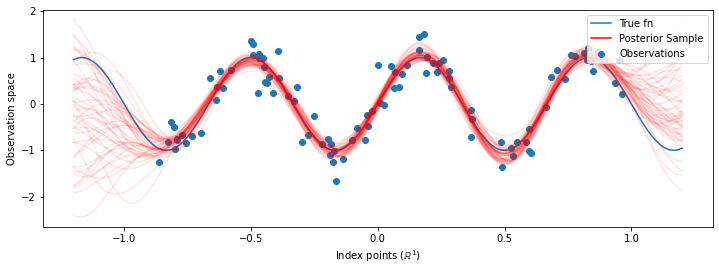
# Plot the true function, observations, and posterior samples.
plt.figure(figsize=(12, 4))
plt.plot(predictive_index_points_, sinusoid(predictive_index_points_),
label='True fn')
plt.scatter(observation_index_points_[:, 0], observations_,
label='Observations')
for i in range(num_samples):
plt.plot(predictive_index_points_, samples[i, :], c='r', alpha=.1,
label='Posterior Sample' if i == 0 else None)
leg = plt.legend(loc='upper right')
for lh in leg.legendHandles:
lh.set_alpha(1)
plt.xlabel(r"Index points ($\mathbb{R}^1$)")
plt.ylabel("Observation space")
plt.show()
Meminggirkan hiperparameter dengan HMC
Daripada mengoptimalkan hyperparameter, mari kita coba mengintegrasikannya dengan Hamiltonian Monte Carlo. Pertama-tama kita akan mendefinisikan dan menjalankan sampler untuk menggambar kira-kira dari distribusi posterior melalui hyperparameter kernel, berdasarkan pengamatan.
num_results = 100
num_burnin_steps = 50
sampler = tfp.mcmc.TransformedTransitionKernel(
tfp.mcmc.NoUTurnSampler(
target_log_prob_fn=target_log_prob,
step_size=tf.cast(0.1, tf.float64)),
bijector=[constrain_positive, constrain_positive, constrain_positive])
adaptive_sampler = tfp.mcmc.DualAveragingStepSizeAdaptation(
inner_kernel=sampler,
num_adaptation_steps=int(0.8 * num_burnin_steps),
target_accept_prob=tf.cast(0.75, tf.float64))
initial_state = [tf.cast(x, tf.float64) for x in [1., 1., 1.]]
# Speed up sampling by tracing with `tf.function`.
@tf.function(autograph=False, jit_compile=False)
def do_sampling():
return tfp.mcmc.sample_chain(
kernel=adaptive_sampler,
current_state=initial_state,
num_results=num_results,
num_burnin_steps=num_burnin_steps,
trace_fn=lambda current_state, kernel_results: kernel_results)
t0 = time.time()
samples, kernel_results = do_sampling()
t1 = time.time()
print("Inference ran in {:.2f}s.".format(t1-t0))
Inference ran in 9.00s.
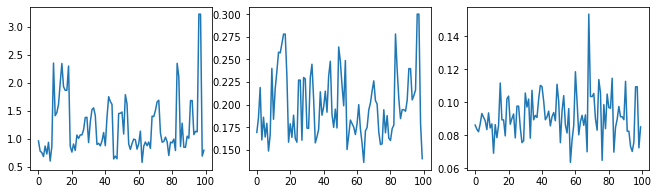
Mari kita periksa kewarasan sampler dengan memeriksa jejak hyperparameter.
(amplitude_samples,
length_scale_samples,
observation_noise_variance_samples) = samples
f = plt.figure(figsize=[15, 3])
for i, s in enumerate(samples):
ax = f.add_subplot(1, len(samples) + 1, i + 1)
ax.plot(s)
Sekarang bukan membangun GP tunggal dengan hyperparameters dioptimalkan, kita membangun distribusi prediktif posterior sebagai campuran dokter, masing-masing didefinisikan oleh sampel dari distribusi posterior lebih hyperparameters. Ini kira-kira terintegrasi dengan parameter posterior melalui pengambilan sampel Monte Carlo untuk menghitung distribusi prediktif marginal di lokasi yang tidak teramati.
# The sampled hyperparams have a leading batch dimension, `[num_results, ...]`,
# so they construct a *batch* of kernels.
batch_of_posterior_kernels = tfk.ExponentiatedQuadratic(
amplitude_samples, length_scale_samples)
# The batch of kernels creates a batch of GP predictive models, one for each
# posterior sample.
batch_gprm = tfd.GaussianProcessRegressionModel(
kernel=batch_of_posterior_kernels,
index_points=predictive_index_points_,
observation_index_points=observation_index_points_,
observations=observations_,
observation_noise_variance=observation_noise_variance_samples,
predictive_noise_variance=0.)
# To construct the marginal predictive distribution, we average with uniform
# weight over the posterior samples.
predictive_gprm = tfd.MixtureSameFamily(
mixture_distribution=tfd.Categorical(logits=tf.zeros([num_results])),
components_distribution=batch_gprm)
num_samples = 50
samples = predictive_gprm.sample(num_samples)
# Plot the true function, observations, and posterior samples.
plt.figure(figsize=(12, 4))
plt.plot(predictive_index_points_, sinusoid(predictive_index_points_),
label='True fn')
plt.scatter(observation_index_points_[:, 0], observations_,
label='Observations')
for i in range(num_samples):
plt.plot(predictive_index_points_, samples[i, :], c='r', alpha=.1,
label='Posterior Sample' if i == 0 else None)
leg = plt.legend(loc='upper right')
for lh in leg.legendHandles:
lh.set_alpha(1)
plt.xlabel(r"Index points ($\mathbb{R}^1$)")
plt.ylabel("Observation space")
plt.show()
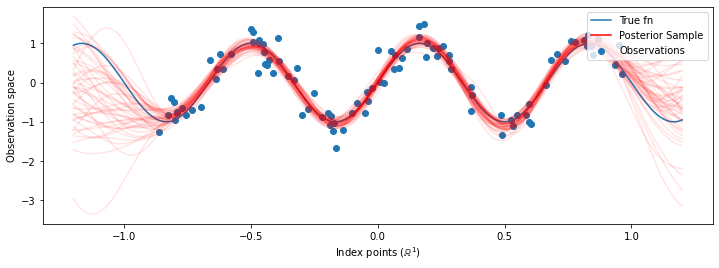
Meskipun perbedaannya tidak kentara dalam kasus ini, secara umum, kami mengharapkan distribusi prediktif posterior untuk digeneralisasi lebih baik (memberikan kemungkinan lebih tinggi untuk data yang ditahan) daripada hanya menggunakan parameter yang paling mungkin seperti yang kami lakukan di atas.
