- الوصف :
"ogbg-molpcba" عبارة عن مجموعة بيانات جزيئية مأخوذة من اختبار PubChem BioAssay. إنها مجموعة بيانات تنبؤ بالرسم البياني من Open Graph Benchmark (OGB).
مجموعة البيانات هذه تجريبية ، و API عرضة للتغيير في الإصدارات المستقبلية.
الوصف أدناه لمجموعة البيانات مقتبس من ورقة OGB:
نمط الإدخال
تتم معالجة جميع الجزيئات مسبقًا باستخدام RDKit ([1]).
- يمثل كل رسم بياني جزيءًا ، حيث العقد عبارة عن ذرات ، والحواف عبارة عن روابط كيميائية.
- ميزات عقدة الإدخال 9 أبعاد ، تحتوي على عدد ذري و chirality ، بالإضافة إلى ميزات ذرية إضافية مثل الشحنة الرسمية وما إذا كانت الذرة في الحلقة.
- ميزات حافة الإدخال ثلاثية الأبعاد ، تحتوي على نوع الرابطة ، والكيمياء الفراغية للسندات ، بالإضافة إلى ميزة الرابطة الإضافية التي تشير إلى ما إذا كانت الرابطة مترافقة.
يتوفر الوصف الدقيق لجميع الميزات على https://github.com/snap-stanford/ogb/blob/master/ogb/utils/features.py
تنبؤ
المهمة هي التنبؤ بـ 128 نشاطًا بيولوجيًا مختلفًا (غير نشط / نشط). انظر [2] و [3] لمزيد من الوصف حول هذه الأهداف. لا تنطبق جميع الأهداف على كل جزيء: فالأهداف المفقودة يشار إليها بواسطة NaNs.
مراجع
[1]: جريج لاندروم وآخرون. "RDKit: معلوماتية chemin مفتوحة المصدر". عنوان URL: https://github.com/rdkit/rdkit
[2]: بهارات رامسوندار ، وستيفن كيرنز ، وباتريك رايلي ، وديل ويبستر ، وديفيد كونيردينج ، وفيجاي باندي. "شبكات متعددة المهام على نطاق واسع لاكتشاف الأدوية". URL: https://arxiv.org/pdf/1502.02072.pdf
[3]: زينكين وو ، وبارات رامسوندار ، وإيفان إن فاينبرغ ، وجوزيف جوميز ، وكالب جينييس ، وآنيش س.بابو ، وكارل ليسوينج ، وفيجاي باندي. MoleculeNet: معيار للتعلم الآلي الجزيئي. العلوم الكيميائية ، 9 (2): 513-530 ، 2018.
الصفحة الرئيسية : https://ogb.stanford.edu/docs/graphprop
كود المصدر :
tfds.datasets.ogbg_molpcba.Builderإصدارات :
-
0.1.0: الإصدار الأولي لواجهة برمجة التطبيقات التجريبية. -
0.1.1: يعرض عدد الحواف في كل رسم بياني بشكل صريح. -
0.1.2: إضافة حقل البيانات الوصفية لـ GraphVisualizer. -
0.1.3(افتراضي): أضف حقل بيانات التعريف لأسماء المهام الفردية.
-
حجم التحميل :
37.70 MiBحجم مجموعة البيانات :
822.53 MiBالتخزين المؤقت التلقائي ( التوثيق ): لا
الانقسامات :
| ينقسم | أمثلة |
|---|---|
'test' | 43793 |
'train' | 350343 |
'validation' | 43793 |
- هيكل الميزة :
FeaturesDict({
'edge_feat': Tensor(shape=(None, 3), dtype=float32),
'edge_index': Tensor(shape=(None, 2), dtype=int64),
'labels': Tensor(shape=(128,), dtype=float32),
'node_feat': Tensor(shape=(None, 9), dtype=float32),
'num_edges': Tensor(shape=(None,), dtype=int64),
'num_nodes': Tensor(shape=(None,), dtype=int64),
})
- وثائق الميزة :
| ميزة | فصل | شكل | نوع | وصف |
|---|---|---|---|---|
| الميزات | ||||
| الحافة | موتر | (لا شيء ، 3) | تعويم 32 | |
| حافة_الفهرس | موتر | (لا شيء ، 2) | int64 | |
| تسميات | موتر | (128 ،) | تعويم 32 | |
| node_feat | موتر | (لا شيء ، 9) | تعويم 32 | |
| عدد_حواف | موتر | (لا أحد،) | int64 | |
| num_nodes | موتر | (لا أحد،) | int64 |
المفاتيح الخاضعة للإشراف (انظر المستند
as_supervised):Noneالشكل ( tfds.show_examples ):
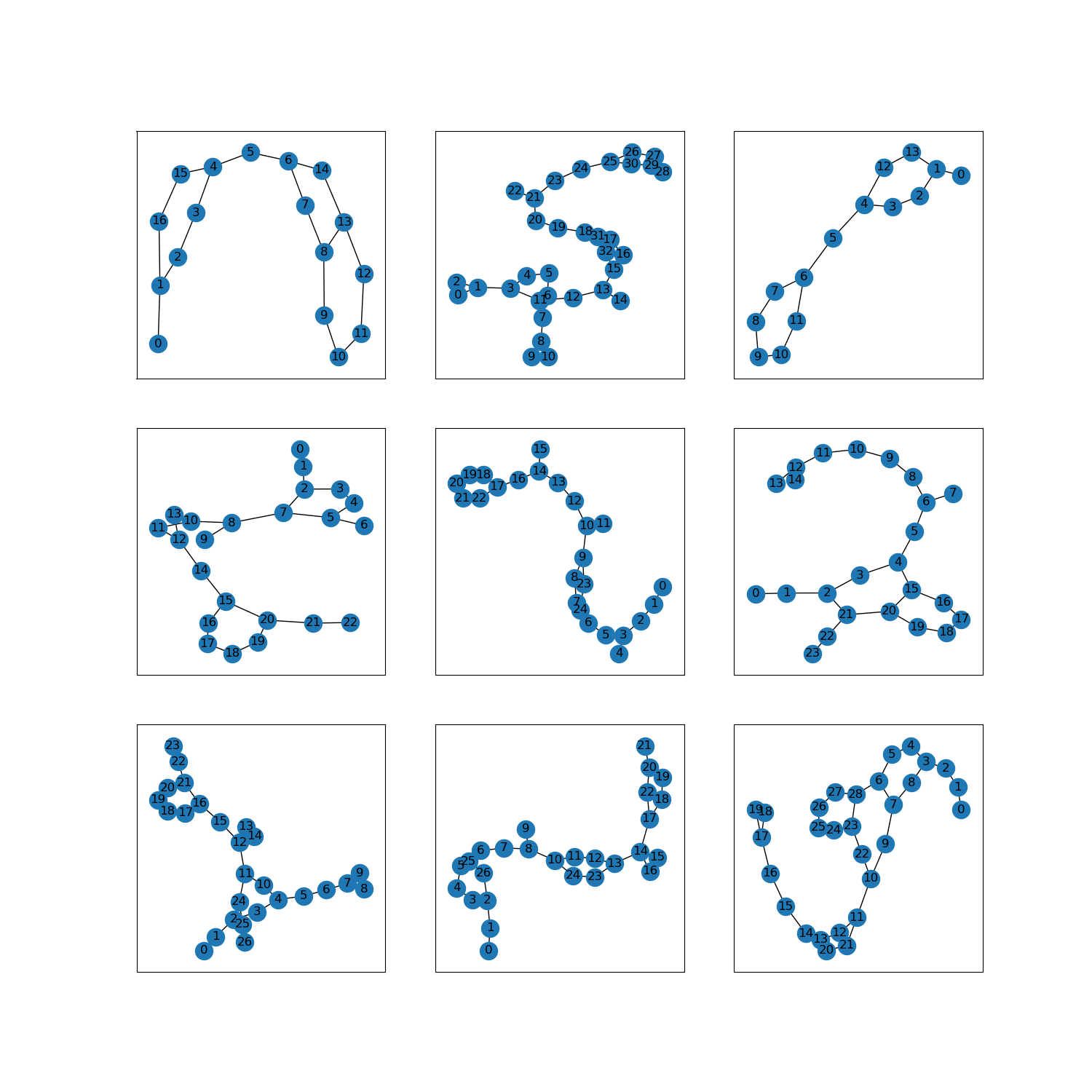
- أمثلة ( tfds.as_dataframe ):
- الاقتباس :
@inproceedings{DBLP:conf/nips/HuFZDRLCL20,
author = {Weihua Hu and
Matthias Fey and
Marinka Zitnik and
Yuxiao Dong and
Hongyu Ren and
Bowen Liu and
Michele Catasta and
Jure Leskovec},
editor = {Hugo Larochelle and
Marc Aurelio Ranzato and
Raia Hadsell and
Maria{-}Florina Balcan and
Hsuan{-}Tien Lin},
title = {Open Graph Benchmark: Datasets for Machine Learning on Graphs},
booktitle = {Advances in Neural Information Processing Systems 33: Annual Conference
on Neural Information Processing Systems 2020, NeurIPS 2020, December
6-12, 2020, virtual},
year = {2020},
url = {https://proceedings.neurips.cc/paper/2020/hash/fb60d411a5c5b72b2e7d3527cfc84fd0-Abstract.html},
timestamp = {Tue, 19 Jan 2021 15:57:06 +0100},
biburl = {https://dblp.org/rec/conf/nips/HuFZDRLCL20.bib},
bibsource = {dblp computer science bibliography, https://dblp.org}
}

