- opis :
„ogbg-molpcba” to zestaw danych molekularnych pobrany z PubChem BioAssay. Jest to zestaw danych do przewidywania wykresów z Open Graph Benchmark (OGB).
Ten zestaw danych jest eksperymentalny, a interfejs API może ulec zmianie w przyszłych wersjach.
Poniższy opis zbioru danych został zaadaptowany z artykułu OGB:
Format wejściowy
Wszystkie cząsteczki są wstępnie przetwarzane przy użyciu RDKit ([1]).
- Każdy wykres przedstawia cząsteczkę, w której węzły to atomy, a krawędzie to wiązania chemiczne.
- Cechy węzła wejściowego są 9-wymiarowe, zawierają liczbę atomową i chiralność, a także inne dodatkowe cechy atomu, takie jak ładunek formalny i to, czy atom jest w pierścieniu.
- Cechy krawędzi wejściowej są trójwymiarowe, zawierają typ wiązania, stereochemię wiązania, a także dodatkową cechę wiązania wskazującą, czy wiązanie jest sprzężone.
Dokładny opis wszystkich funkcji dostępny jest pod adresem https://github.com/snap-stanford/ogb/blob/master/ogb/utils/features.py
Prognoza
Zadanie polega na przewidzeniu 128 różnych aktywności biologicznych (nieaktywne/aktywne). Więcej informacji na temat tych celów można znaleźć w [2] i [3]. Nie wszystkie cele dotyczą każdej cząsteczki: brakujące cele są oznaczone NaN.
Bibliografia
[1]: Greg Landrum i in. „RDKit: cheminformatyka typu open source”. URL: https://github.com/rdkit/rdkit
[2]: Bharath Ramsundar, Steven Kearnes, Patrick Riley, Dale Webster, David Konerding i Vijay Pande. „Masowo wielozadaniowe sieci do odkrywania leków”. URL: https://arxiv.org/pdf/1502.02072.pdf
[3]: Zhenqin Wu, Bharath Ramsundar, Evan N. Feinberg, Joseph Gomes, Caleb Geniesse, Aneesh S. Pappu, Karl Leswing i Vijay Pande. MoleculeNet: punkt odniesienia dla molekularnego uczenia maszynowego. Chemical Science, 9(2):513-530, 2018.
Strona główna : https://ogb.stanford.edu/docs/graphprop
Kod źródłowy :
tfds.datasets.ogbg_molpcba.BuilderWersje :
-
0.1.0: Pierwsza wersja eksperymentalnego interfejsu API. -
0.1.1: Wyraźnie ujawnia liczbę krawędzi na każdym wykresie. -
0.1.2: Dodaj pole metadanych dla GraphVisualizer. -
0.1.3(domyślnie): Dodaj pole metadanych dla nazw poszczególnych zadań.
-
Rozmiar pliku do pobrania :
37.70 MiBRozmiar zbioru danych :
822.53 MiBAutomatyczne buforowanie ( dokumentacja ): Nie
Podziały :
| Podział | Przykłady |
|---|---|
'test' | 43793 |
'train' | 350343 |
'validation' | 43793 |
- Struktura funkcji :
FeaturesDict({
'edge_feat': Tensor(shape=(None, 3), dtype=float32),
'edge_index': Tensor(shape=(None, 2), dtype=int64),
'labels': Tensor(shape=(128,), dtype=float32),
'node_feat': Tensor(shape=(None, 9), dtype=float32),
'num_edges': Tensor(shape=(None,), dtype=int64),
'num_nodes': Tensor(shape=(None,), dtype=int64),
})
- Dokumentacja funkcji :
| Funkcja | Klasa | Kształt | Typ D | Opis |
|---|---|---|---|---|
| FunkcjeDict | ||||
| krawędź_feat | Napinacz | (Brak, 3) | pływak32 | |
| indeks_krawędzi | Napinacz | (Brak, 2) | int64 | |
| etykiety | Napinacz | (128,) | pływak32 | |
| node_feat | Napinacz | (Brak, 9) | pływak32 | |
| liczba_krawędzi | Napinacz | (Nic,) | int64 | |
| liczba_węzłów | Napinacz | (Nic,) | int64 |
Klucze nadzorowane (Zobacz dokument
as_supervised):NoneRysunek ( tfds.show_examples ):
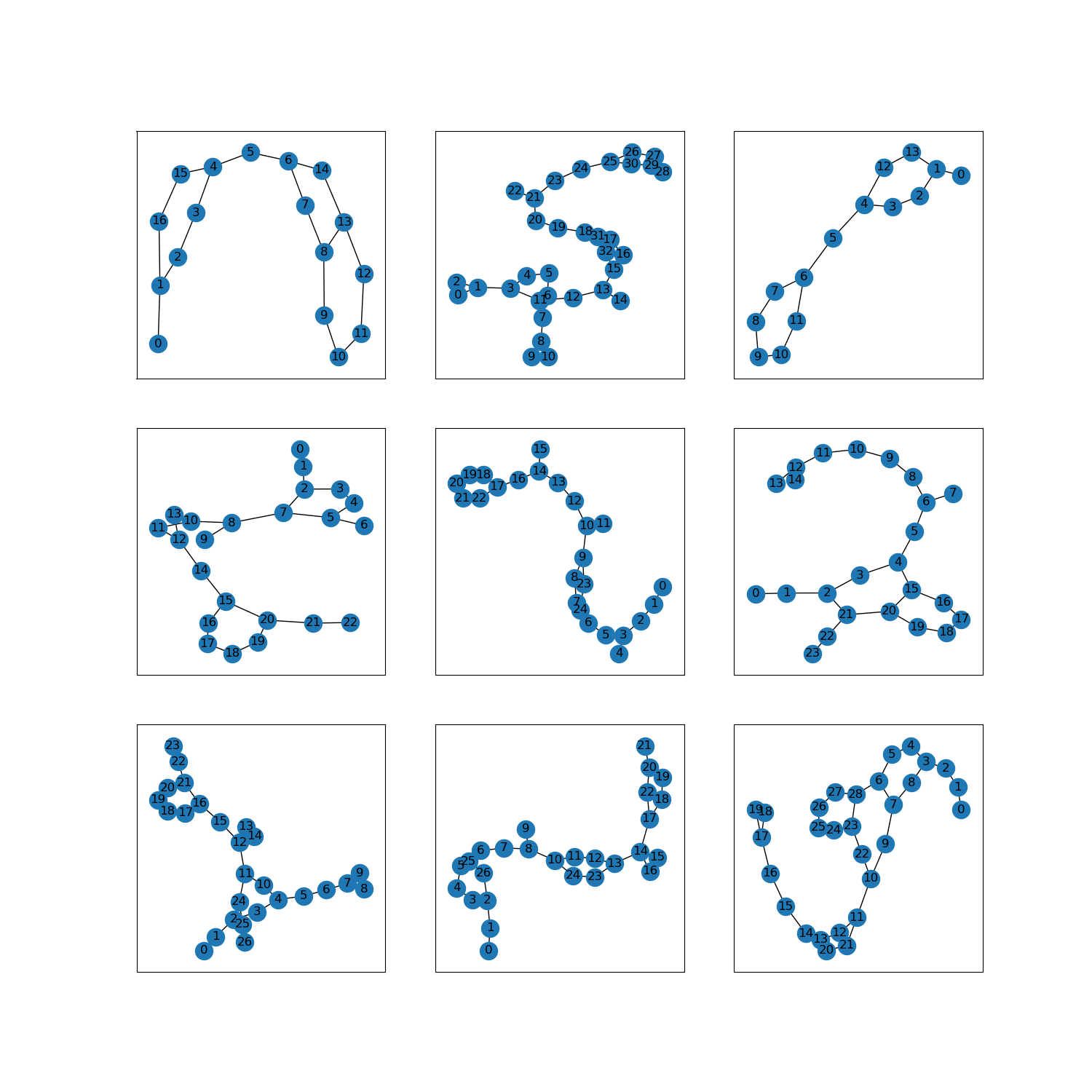
- Przykłady ( tfds.as_dataframe ):
- Cytat :
@inproceedings{DBLP:conf/nips/HuFZDRLCL20,
author = {Weihua Hu and
Matthias Fey and
Marinka Zitnik and
Yuxiao Dong and
Hongyu Ren and
Bowen Liu and
Michele Catasta and
Jure Leskovec},
editor = {Hugo Larochelle and
Marc Aurelio Ranzato and
Raia Hadsell and
Maria{-}Florina Balcan and
Hsuan{-}Tien Lin},
title = {Open Graph Benchmark: Datasets for Machine Learning on Graphs},
booktitle = {Advances in Neural Information Processing Systems 33: Annual Conference
on Neural Information Processing Systems 2020, NeurIPS 2020, December
6-12, 2020, virtual},
year = {2020},
url = {https://proceedings.neurips.cc/paper/2020/hash/fb60d411a5c5b72b2e7d3527cfc84fd0-Abstract.html},
timestamp = {Tue, 19 Jan 2021 15:57:06 +0100},
biburl = {https://dblp.org/rec/conf/nips/HuFZDRLCL20.bib},
bibsource = {dblp computer science bibliography, https://dblp.org}
}

