- Descripción :
El punto de referencia PatchCamelyon es un conjunto de datos de clasificación de imágenes nuevo y desafiante. Consta de 327.680 imágenes en color (96 x 96 px) extraídas de exploraciones histopatológicas de secciones de ganglios linfáticos. Cada imagen está marcada con una etiqueta binaria que indica la presencia de tejido metastásico. PCam proporciona un nuevo punto de referencia para los modelos de aprendizaje automático: más grande que CIFAR10, más pequeño que Imagenet y entrenable en una sola GPU.
Documentación adicional : Explorar en artículos con código
Página de inicio : https://patchcamelyon.grand-challenge.org/
Código fuente :
tfds.datasets.patch_camelyon.BuilderVersiones :
-
2.0.0(predeterminado): Nueva API dividida ( https://tensorflow.org/datasets/splits )
-
Tamaño de descarga :
7.48 GiBTamaño del conjunto de datos :
7.06 GiBAlmacenamiento en caché automático ( documentación ): No
Divisiones :
| Dividir | Ejemplos |
|---|---|
'test' | 32.768 |
'train' | 262,144 |
'validation' | 32.768 |
- Estructura de características :
FeaturesDict({
'id': Text(shape=(), dtype=string),
'image': Image(shape=(96, 96, 3), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentación de funciones :
| Característica | Clase | Forma | tipo D | Descripción |
|---|---|---|---|---|
| FuncionesDict | ||||
| identificación | Texto | cadena | ||
| imagen | Imagen | (96, 96, 3) | uint8 | |
| etiqueta | Etiqueta de clase | int64 |
Claves supervisadas (Ver documento
as_supervised):('image', 'label')Figura ( tfds.show_examples ):
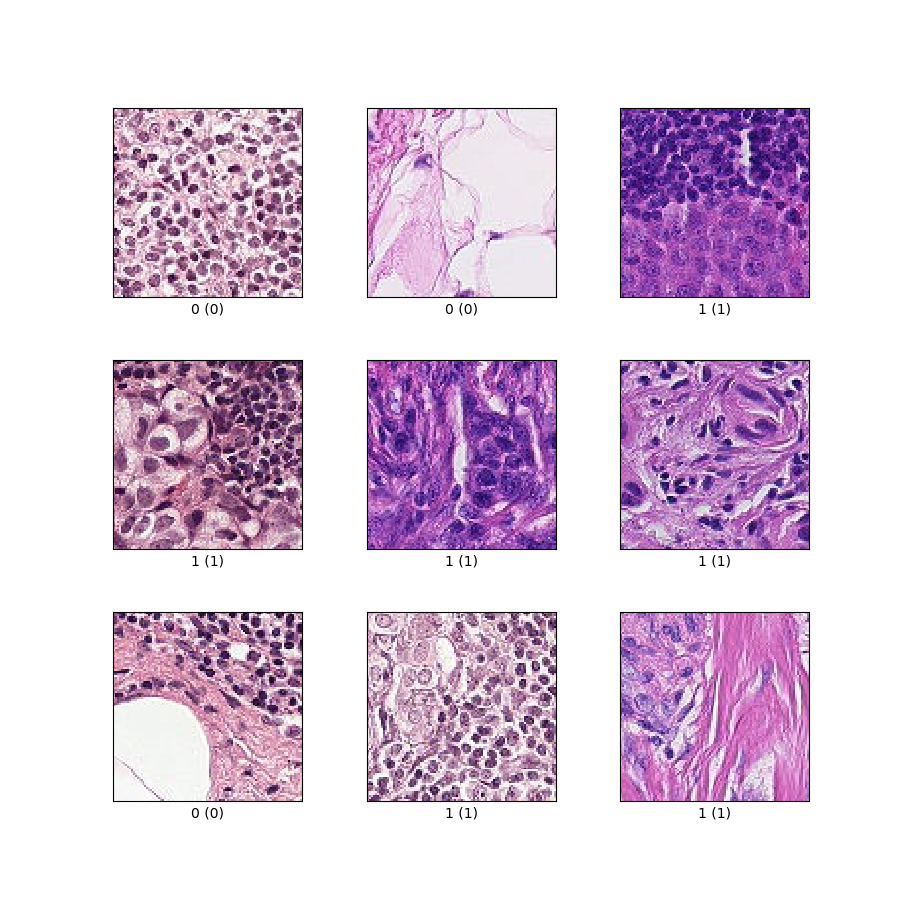
- Ejemplos ( tfds.as_dataframe ):
- Citación :
@misc{b_s_veeling_j_linmans_j_winkens_t_cohen_2018_2546921,
author = {B. S. Veeling, J. Linmans, J. Winkens, T. Cohen, M. Welling},
title = {Rotation Equivariant CNNs for Digital Pathology},
month = sep,
year = 2018,
doi = {10.1007/978-3-030-00934-2_24},
url = {https://doi.org/10.1007/978-3-030-00934-2_24}
}

